[PYTHON] Pandas Benutzerhandbuch "Tabellenformatierung und Pivot-Tabelle" (offizielles Dokument Japanische Übersetzung)
Dieser Artikel ist eine teilweise maschinelle Übersetzung der offiziellen Pandas-Dokumentation Benutzerhandbuch - Umformen und Pivot-Tabellen. Es ist eine Modifikation eines unnatürlichen Satzes.
Wenn Sie falsche Übersetzungen, alternative Übersetzungen, Fragen usw. haben, verwenden Sie bitte den Kommentarbereich oder bearbeiten Sie die Anfrage.
Tischformung und Drehtisch
Formatierung durch Schwenken eines DataFrame-Objekts
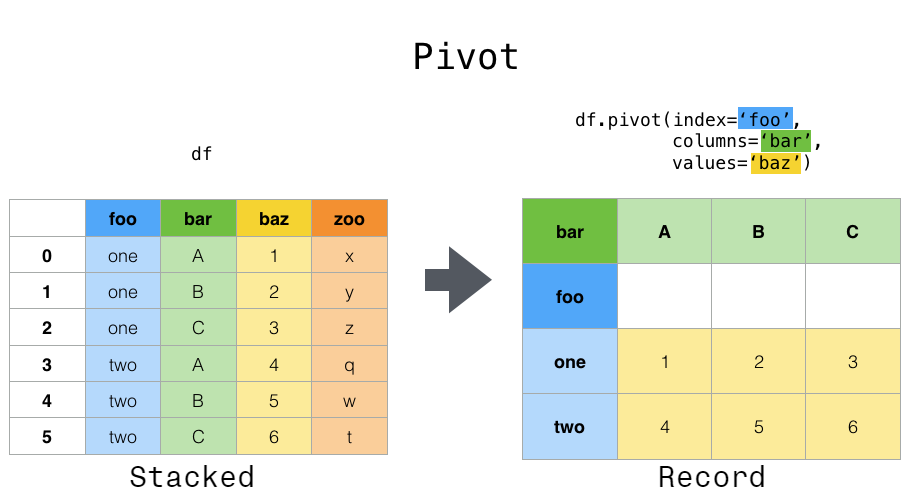
Daten werden häufig in sogenannten "Stacks" oder "Records" gespeichert.
In [1]: df
Out[1]:
date variable value
0 2000-01-03 A 0.469112
1 2000-01-04 A -0.282863
2 2000-01-05 A -1.509059
3 2000-01-03 B -1.135632
4 2000-01-04 B 1.212112
5 2000-01-05 B -0.173215
6 2000-01-03 C 0.119209
7 2000-01-04 C -1.044236
8 2000-01-05 C -0.861849
9 2000-01-03 D -2.104569
10 2000-01-04 D -0.494929
11 2000-01-05 D 1.071804
Übrigens, wie man den obigen "DataFrame" erstellt, ist wie folgt.
import pandas._testing as tm
def unpivot(frame):
N, K = frame.shape
data = {'value': frame.to_numpy().ravel('F'),
'variable': np.asarray(frame.columns).repeat(N),
'date': np.tile(np.asarray(frame.index), K)}
return pd.DataFrame(data, columns=['date', 'variable', 'value'])
df = unpivot(tm.makeTimeDataFrame(3))
So wählen Sie alle Zeilen mit der Variablen "A" aus:
In [2]: df[df['variable'] == 'A']
Out[2]:
date variable value
0 2000-01-03 A 0.469112
1 2000-01-04 A -0.282863
2 2000-01-05 A -1.509059
Angenommen, Sie möchten diese Variablen verwenden, um Zeitreihenoperationen auszuführen. In diesem Fall wäre es besser, die einzelnen Beobachtungen durch eine eindeutige Variable "Spalten" und einen Datumsindex zu identifizieren. Um die Daten in dieses Format umzuformen, DataFrame.pivot () Verwenden Sie die Methode .DataFrame.pivot) (Funktion der obersten Ebene pivot (). .pivot) ist ebenfalls implementiert).
In [3]: df.pivot(index='date', columns='variable', values='value')
Out[3]:
variable A B C D
date
2000-01-03 0.469112 -1.135632 0.119209 -2.104569
2000-01-04 -0.282863 1.212112 -1.044236 -0.494929
2000-01-05 -1.509059 -0.173215 -0.861849 1.071804
Wenn das Argument "values" weggelassen wird und die Eingabe "DataFrame" mehrere Wertespalten enthält, die "pivot" nicht als Spalten oder Indizes zugewiesen werden, hat der resultierende "Pivot" "DataFrame" die höchste Ebene jeder Wertespalte. Verfügt über eine hierarchische Spalte (https://qiita.com/nkay/items/ Multi-Index Advanced Index), die angibt.
In [4]: df['value2'] = df['value'] * 2
In [5]: pivoted = df.pivot(index='date', columns='variable')
In [6]: pivoted
Out[6]:
value ... value2
variable A B C ... B C D
date ...
2000-01-03 0.469112 -1.135632 0.119209 ... -2.271265 0.238417 -4.209138
2000-01-04 -0.282863 1.212112 -1.044236 ... 2.424224 -2.088472 -0.989859
2000-01-05 -1.509059 -0.173215 -0.861849 ... -0.346429 -1.723698 2.143608
[3 rows x 8 columns]
Sie können dann eine Teilmenge aus dem geschwenkten "DataFrame" auswählen.
In [7]: pivoted['value2']
Out[7]:
variable A B C D
date
2000-01-03 0.938225 -2.271265 0.238417 -4.209138
2000-01-04 -0.565727 2.424224 -2.088472 -0.989859
2000-01-05 -3.018117 -0.346429 -1.723698 2.143608
Beachten Sie, dass wenn die Daten vom gleichen Typ sind, eine Ansicht der zugrunde liegenden Daten zurückgegeben wird.
: ballot_box_with_check: ** Hinweis ** Wenn das Index / Spalten-Paar nicht eindeutig ist, wird
pivot ()Löst den Fehler "ValueError: Index enthält doppelte Einträge, kann nicht umgeformt werden" aus. In diesem Fall sollten Siepivot_table ()verwenden. Bitte gib mir. Dies ist ein verallgemeinerter Pivot, der doppelte Werte für ein einzelnes Index / Spalten-Paar verarbeiten kann.
Formänderung durch Stapeln und Entstapeln
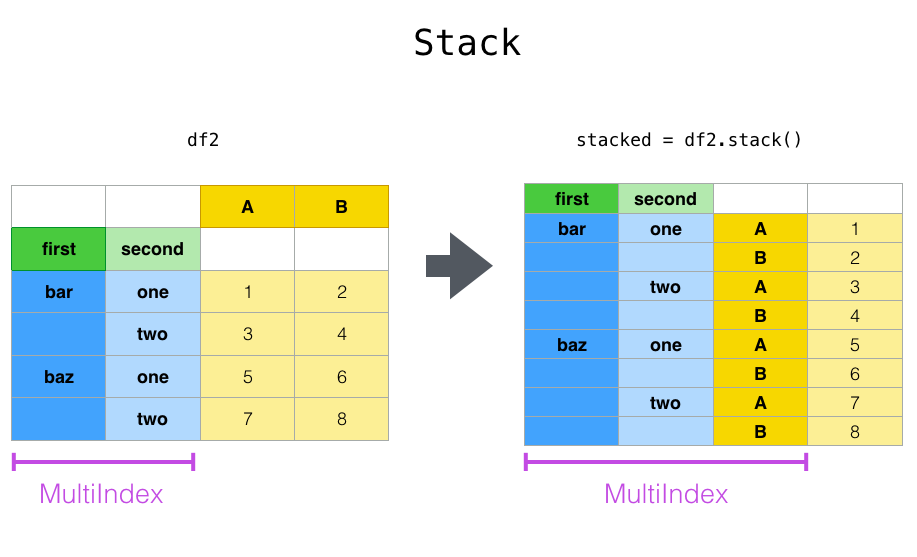
pivot () Dieselbe Serie, die eng mit der Methode verwandt ist Stack () verfügbar in Series und DataFrame für die Methode von # pandas.DataFrame.stack) und unstack () Es gibt eine Methode. Diese Methoden funktionieren mit dem Objekt "MultiIndex" (siehe Kapitel über hierarchische Indizes (https://qiita.com/nkay/items/Multi-Index Advanced Indexes)). Die Grundfunktionen dieser Methoden sind wie folgt:
--stack: "Pivots" die Ebene der (möglicherweise hierarchischen) Spaltenbezeichnung und gibt einen neuen DataFrame mit dieser Zeilenbezeichnung auf der innersten Ebene des Index zurück.
--unstack: (umgekehrte Stapelung) (wahrscheinlich hierarchisch) Die Zeilenindexebene wird um die Spaltenachse "geschwenkt", um einen rekonstruierten "DataFrame" mit der Spaltenbezeichnung der innersten Ebene zu generieren Ich werde.
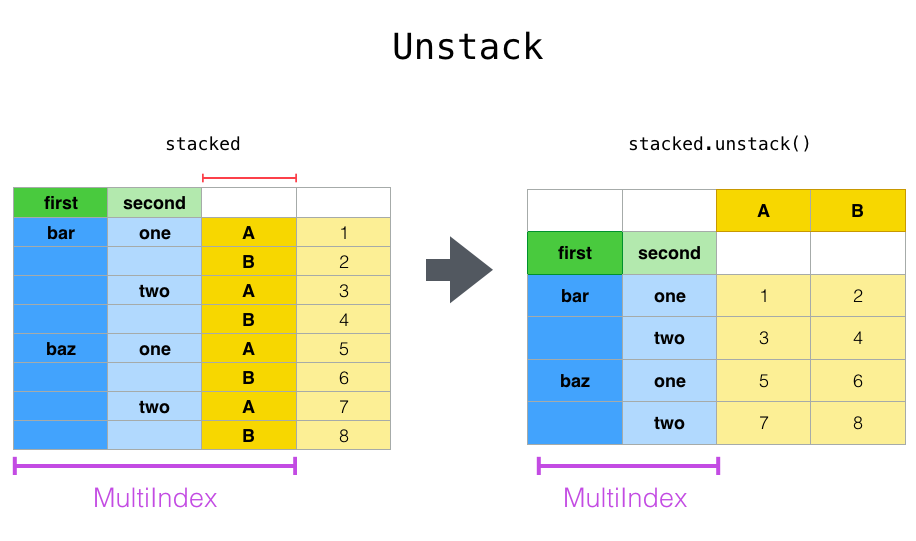
Es ist einfacher zu verstehen, wenn Sie sich ein aktuelles Beispiel ansehen. Es handelt sich um denselben Datensatz, den wir im Kapitel Hierarchischer Index gesehen haben.
In [8]: tuples = list(zip(*[['bar', 'bar', 'baz', 'baz',
...: 'foo', 'foo', 'qux', 'qux'],
...: ['one', 'two', 'one', 'two',
...: 'one', 'two', 'one', 'two']]))
...:
In [9]: index = pd.MultiIndex.from_tuples(tuples, names=['first', 'second'])
In [10]: df = pd.DataFrame(np.random.randn(8, 2), index=index, columns=['A', 'B'])
In [11]: df2 = df[:4]
In [12]: df2
Out[12]:
A B
first second
bar one 0.721555 -0.706771
two -1.039575 0.271860
baz one -0.424972 0.567020
two 0.276232 -1.087401
Die Funktion "Stapel" "komprimiert" die Ebene der Spalte "DataFrame", um eine der folgenden Eigenschaften zu erzeugen:
--Series: Für einfache Spaltenindizes.
--DataFrame: Wenn die Spalte "MultiIndex" enthält.
Wenn die Spalte "MultiIndex" enthält, können Sie die zu stapelnde Ebene auswählen. Die gestapelte Ebene ist die niedrigste Ebene der neuen Spalte "MultiIndex".
In [13]: stacked = df2.stack()
In [14]: stacked
Out[14]:
first second
bar one A 0.721555
B -0.706771
two A -1.039575
B 0.271860
baz one A -0.424972
B 0.567020
two A 0.276232
B -1.087401
dtype: float64
Verwenden Sie für einen "gestapelten" "DataFrame" oder "Series" (dh "Index" ist "MultiIndex") "Unstack", um das Gegenteil von "Stack" auszuführen. Standardmäßig wird die ** niedrigste Ebene ** entstapelt.
In [15]: stacked.unstack()
Out[15]:
A B
first second
bar one 0.721555 -0.706771
two -1.039575 0.271860
baz one -0.424972 0.567020
two 0.276232 -1.087401
In [16]: stacked.unstack(1)
Out[16]:
second one two
first
bar A 0.721555 -1.039575
B -0.706771 0.271860
baz A -0.424972 0.276232
B 0.567020 -1.087401
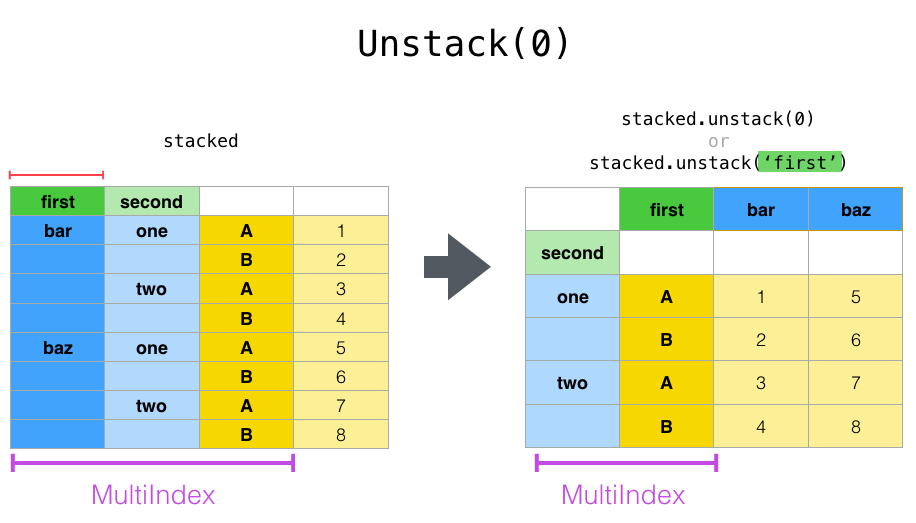
In [17]: stacked.unstack(0)
Out[17]:
first bar baz
second
one A 0.721555 -0.424972
B -0.706771 0.567020
two A -1.039575 0.276232
B 0.271860 -1.087401
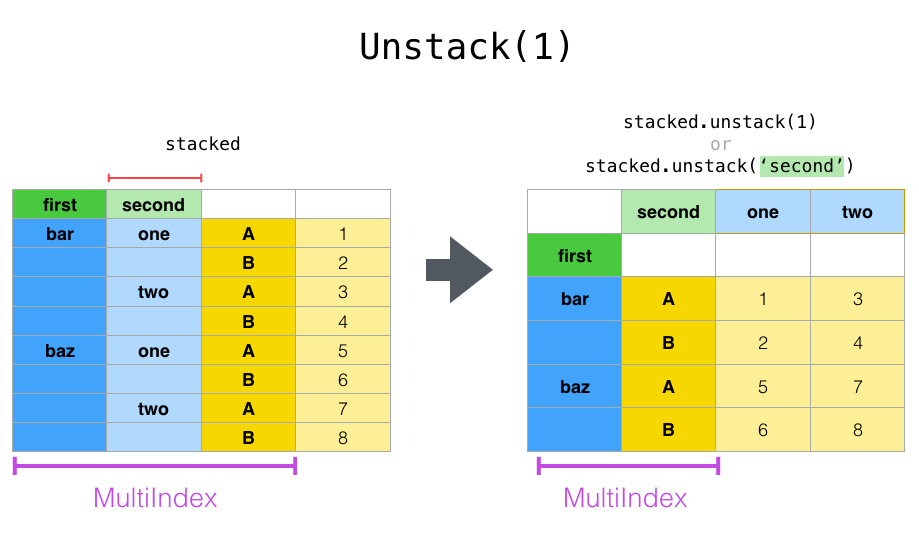
Wenn der Index einen Namen hat, können Sie den Ebenennamen verwenden, anstatt die Ebenennummer anzugeben.
In [18]: stacked.unstack('second')
Out[18]:
second one two
first
bar A 0.721555 -1.039575
B -0.706771 0.271860
baz A -0.424972 0.276232
B 0.567020 -1.087401
Beachten Sie, dass die Methoden stack und unstack implizit die zugehörigen Indexebenen sortieren. Wenn Sie also "stapeln" und dann "entstapeln" (oder umgekehrt), erhalten Sie eine ** sortierte ** Kopie des ursprünglichen "DataFrame" oder "Series".
In [19]: index = pd.MultiIndex.from_product([[2, 1], ['a', 'b']])
In [20]: df = pd.DataFrame(np.random.randn(4), index=index, columns=['A'])
In [21]: df
Out[21]:
A
2 a -0.370647
b -1.157892
1 a -1.344312
b 0.844885
In [22]: all(df.unstack().stack() == df.sort_index())
Out[22]: True
Der obige Code löst einen "TypeError" aus, wenn der Aufruf von "sort_index" entfernt wird.
Mehrere Ebenen
Sie können auch mehrere Ebenen gleichzeitig stapeln oder entstapeln, indem Sie eine Liste von Ebenen übergeben. In diesem Fall ist das Endergebnis das gleiche, als ob jede Ebene in der Liste einzeln verarbeitet worden wäre.
In [23]: columns = pd.MultiIndex.from_tuples([
....: ('A', 'cat', 'long'), ('B', 'cat', 'long'),
....: ('A', 'dog', 'short'), ('B', 'dog', 'short')],
....: names=['exp', 'animal', 'hair_length']
....: )
....:
In [24]: df = pd.DataFrame(np.random.randn(4, 4), columns=columns)
In [25]: df
Out[25]:
exp A B A B
animal cat cat dog dog
hair_length long long short short
0 1.075770 -0.109050 1.643563 -1.469388
1 0.357021 -0.674600 -1.776904 -0.968914
2 -1.294524 0.413738 0.276662 -0.472035
3 -0.013960 -0.362543 -0.006154 -0.923061
In [26]: df.stack(level=['animal', 'hair_length'])
Out[26]:
exp A B
animal hair_length
0 cat long 1.075770 -0.109050
dog short 1.643563 -1.469388
1 cat long 0.357021 -0.674600
dog short -1.776904 -0.968914
2 cat long -1.294524 0.413738
dog short 0.276662 -0.472035
3 cat long -0.013960 -0.362543
dog short -0.006154 -0.923061
Die Liste der Ebenen kann entweder Ebenennamen oder Ebenennummern enthalten (obwohl die beiden nicht gemischt werden können).
# df.stack(level=['animal', 'hair_length'])
#Dieser Code ist gleich
In [27]: df.stack(level=[1, 2])
Out[27]:
exp A B
animal hair_length
0 cat long 1.075770 -0.109050
dog short 1.643563 -1.469388
1 cat long 0.357021 -0.674600
dog short -1.776904 -0.968914
2 cat long -1.294524 0.413738
dog short 0.276662 -0.472035
3 cat long -0.013960 -0.362543
dog short -0.006154 -0.923061
Fehlende Daten
Diese Funktionen sind auch flexibel im Umgang mit fehlenden Daten und funktionieren auch dann, wenn nicht jede Untergruppe im hierarchischen Index denselben Beschriftungssatz hat. Sie können auch unsortierte Indizes verarbeiten (natürlich können Sie sortieren, indem Sie sort_index aufrufen). Hier ist ein komplexeres Beispiel:
In [28]: columns = pd.MultiIndex.from_tuples([('A', 'cat'), ('B', 'dog'),
....: ('B', 'cat'), ('A', 'dog')],
....: names=['exp', 'animal'])
....:
In [29]: index = pd.MultiIndex.from_product([('bar', 'baz', 'foo', 'qux'),
....: ('one', 'two')],
....: names=['first', 'second'])
....:
In [30]: df = pd.DataFrame(np.random.randn(8, 4), index=index, columns=columns)
In [31]: df2 = df.iloc[[0, 1, 2, 4, 5, 7]]
In [32]: df2
Out[32]:
exp A B A
animal cat dog cat dog
first second
bar one 0.895717 0.805244 -1.206412 2.565646
two 1.431256 1.340309 -1.170299 -0.226169
baz one 0.410835 0.813850 0.132003 -0.827317
foo one -1.413681 1.607920 1.024180 0.569605
two 0.875906 -2.211372 0.974466 -2.006747
qux two -1.226825 0.769804 -1.281247 -0.727707
Wie bereits erwähnt, können Sie mit dem Argument "level" "stack" aufrufen, um auszuwählen, welche Ebene in der Spalte gestapelt werden soll.
In [33]: df2.stack('exp')
Out[33]:
animal cat dog
first second exp
bar one A 0.895717 2.565646
B -1.206412 0.805244
two A 1.431256 -0.226169
B -1.170299 1.340309
baz one A 0.410835 -0.827317
B 0.132003 0.813850
foo one A -1.413681 0.569605
B 1.024180 1.607920
two A 0.875906 -2.006747
B 0.974466 -2.211372
qux two A -1.226825 -0.727707
B -1.281247 0.769804
In [34]: df2.stack('animal')
Out[34]:
exp A B
first second animal
bar one cat 0.895717 -1.206412
dog 2.565646 0.805244
two cat 1.431256 -1.170299
dog -0.226169 1.340309
baz one cat 0.410835 0.132003
dog -0.827317 0.813850
foo one cat -1.413681 1.024180
dog 0.569605 1.607920
two cat 0.875906 0.974466
dog -2.006747 -2.211372
qux two cat -1.226825 -1.281247
dog -0.727707 0.769804
Wenn die Untergruppen nicht über dieselben Beschriftungen verfügen, kann das Entstapeln zu fehlenden Werten führen. Standardmäßig werden fehlende Werte durch die Standardwerte zum Ausfüllen der Lücke für diesen Datentyp ersetzt (z. B. "NaN" für float, "NaT" für datetime-like). Bei ganzzahligen Typen werden die Daten standardmäßig in float konvertiert und der fehlende Wert auf "NaN" gesetzt.
In [35]: df3 = df.iloc[[0, 1, 4, 7], [1, 2]]
In [36]: df3
Out[36]:
exp B
animal dog cat
first second
bar one 0.805244 -1.206412
two 1.340309 -1.170299
foo one 1.607920 1.024180
qux two 0.769804 -1.281247
In [37]: df3.unstack()
Out[37]:
exp B
animal dog cat
second one two one two
first
bar 0.805244 1.340309 -1.206412 -1.170299
foo 1.607920 NaN 1.024180 NaN
qux NaN 0.769804 NaN -1.281247
unstack kann auch ein optionales fill_value-Argument verwenden, das den Wert der fehlenden Daten angibt.
In [38]: df3.unstack(fill_value=-1e9)
Out[38]:
exp B
animal dog cat
second one two one two
first
bar 8.052440e-01 1.340309e+00 -1.206412e+00 -1.170299e+00
foo 1.607920e+00 -1.000000e+09 1.024180e+00 -1.000000e+09
qux -1.000000e+09 7.698036e-01 -1.000000e+09 -1.281247e+00
Für MultiIndex
Selbst wenn die Spalte "MultiIndex" ist, kann sie problemlos entstapelt werden.
In [39]: df[:3].unstack(0)
Out[39]:
exp A B ... A
animal cat dog ... cat dog
first bar baz bar ... baz bar baz
second ...
one 0.895717 0.410835 0.805244 ... 0.132003 2.565646 -0.827317
two 1.431256 NaN 1.340309 ... NaN -0.226169 NaN
[2 rows x 8 columns]
In [40]: df2.unstack(1)
Out[40]:
exp A B ... A
animal cat dog ... cat dog
second one two one ... two one two
first ...
bar 0.895717 1.431256 0.805244 ... -1.170299 2.565646 -0.226169
baz 0.410835 NaN 0.813850 ... NaN -0.827317 NaN
foo -1.413681 0.875906 1.607920 ... 0.974466 0.569605 -2.006747
qux NaN -1.226825 NaN ... -1.281247 NaN -0.727707
[4 rows x 8 columns]
Formen durch Schmelzen
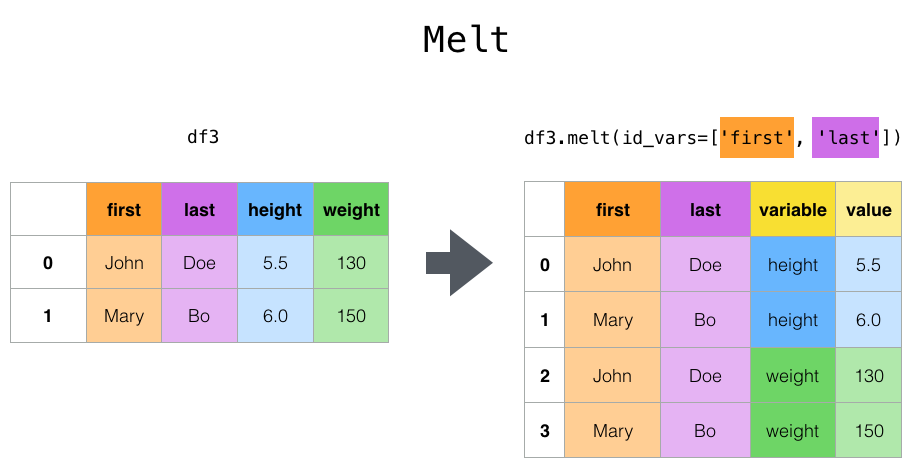
Funktion der obersten Ebene blend () und der zugehörige DataFrame. Schmelze () hat einen oder mehrere DataFrame Wird in einen "Ampivot" -Zustand konvertiert, in dem die Spalten von als * Identifikationsvariablen * und alle anderen Spalten als * Messvariablen * betrachtet werden, wobei nur zwei nicht identifizierte Spalten übrig bleiben, "Variable" und "Wert". Nützlich zum. Die Namen der verbleibenden Spalten können durch Angabe der Parameter "var_name" und "value_name" angepasst werden.
Zum Beispiel
In [41]: cheese = pd.DataFrame({'first': ['John', 'Mary'],
....: 'last': ['Doe', 'Bo'],
....: 'height': [5.5, 6.0],
....: 'weight': [130, 150]})
....:
In [42]: cheese
Out[42]:
first last height weight
0 John Doe 5.5 130
1 Mary Bo 6.0 150
In [43]: cheese.melt(id_vars=['first', 'last'])
Out[43]:
first last variable value
0 John Doe height 5.5
1 Mary Bo height 6.0
2 John Doe weight 130.0
3 Mary Bo weight 150.0
In [44]: cheese.melt(id_vars=['first', 'last'], var_name='quantity')
Out[44]:
first last quantity value
0 John Doe height 5.5
1 Mary Bo height 6.0
2 John Doe weight 130.0
3 Mary Bo weight 150.0
Alternativ ist wide_to_long () eine bequeme Möglichkeit, Paneldaten zu konvertieren. Es ist eine Funktion. Weniger flexibel als blend (), aber mehr Benutzer Es ist freundlich.
In [45]: dft = pd.DataFrame({"A1970": {0: "a", 1: "b", 2: "c"},
....: "A1980": {0: "d", 1: "e", 2: "f"},
....: "B1970": {0: 2.5, 1: 1.2, 2: .7},
....: "B1980": {0: 3.2, 1: 1.3, 2: .1},
....: "X": dict(zip(range(3), np.random.randn(3)))
....: })
....:
In [46]: dft["id"] = dft.index
In [47]: dft
Out[47]:
A1970 A1980 B1970 B1980 X id
0 a d 2.5 3.2 -0.121306 0
1 b e 1.2 1.3 -0.097883 1
2 c f 0.7 0.1 0.695775 2
In [48]: pd.wide_to_long(dft, ["A", "B"], i="id", j="year")
Out[48]:
X A B
id year
0 1970 -0.121306 a 2.5
1 1970 -0.097883 b 1.2
2 1970 0.695775 c 0.7
0 1980 -0.121306 d 3.2
1 1980 -0.097883 e 1.3
2 1980 0.695775 f 0.1
Kombination mit Statistik und GroupBy
Sie sollten sich bewusst sein, dass die Kombination von "Pivot", "Stack" und "Unstack" mit GroupBy und grundlegenden statistischen Funktionen für Serien und DataFrame eine sehr aussagekräftige und schnelle Datenmanipulation sein kann.
In [49]: df
Out[49]:
exp A B A
animal cat dog cat dog
first second
bar one 0.895717 0.805244 -1.206412 2.565646
two 1.431256 1.340309 -1.170299 -0.226169
baz one 0.410835 0.813850 0.132003 -0.827317
two -0.076467 -1.187678 1.130127 -1.436737
foo one -1.413681 1.607920 1.024180 0.569605
two 0.875906 -2.211372 0.974466 -2.006747
qux one -0.410001 -0.078638 0.545952 -1.219217
two -1.226825 0.769804 -1.281247 -0.727707
In [50]: df.stack().mean(1).unstack()
Out[50]:
animal cat dog
first second
bar one -0.155347 1.685445
two 0.130479 0.557070
baz one 0.271419 -0.006733
two 0.526830 -1.312207
foo one -0.194750 1.088763
two 0.925186 -2.109060
qux one 0.067976 -0.648927
two -1.254036 0.021048
#Ähnliche Ergebnisse auf andere Weise
In [51]: df.groupby(level=1, axis=1).mean()
Out[51]:
animal cat dog
first second
bar one -0.155347 1.685445
two 0.130479 0.557070
baz one 0.271419 -0.006733
two 0.526830 -1.312207
foo one -0.194750 1.088763
two 0.925186 -2.109060
qux one 0.067976 -0.648927
two -1.254036 0.021048
In [52]: df.stack().groupby(level=1).mean()
Out[52]:
exp A B
second
one 0.071448 0.455513
two -0.424186 -0.204486
In [53]: df.mean().unstack(0)
Out[53]:
exp A B
animal
cat 0.060843 0.018596
dog -0.413580 0.232430
Schwenktisch
pivot () hat verschiedene Datentypen (Zeichenketten) , Numerischer Wert usw.), aber Pandas bietet pivot_table () zum Schwenken durch Aggregieren numerischer Daten. api / pandas.pivot_table.html # pandas.pivot_table) wird ebenfalls bereitgestellt.
Pivot-Tabelle im Tabellenkalkulationsstil mit der Funktion pivot_table () Kann erstellt werden. Weitere Informationen finden Sie unter Kochbuch.
Es braucht einige Argumente.
--data: DataFrame-Objekt.
--value: Eine Spalte oder Liste von zu aggregierenden Spalten.
--index: Column, Grouper, Array mit der gleichen Länge wie die Daten oder eine Liste davon. Schlüssel zum Gruppieren nach Index in der Pivot-Tabelle. Wenn ein Array übergeben wird, wird es auf die gleiche Weise wie die Spaltenwerte aggregiert.
--columns: Columns, Grouper, Arrays mit der gleichen Länge wie die Daten oder eine Liste davon. Schlüssel zum Gruppieren nach Spalten in der Pivot-Tabelle. Wenn ein Array übergeben wird, wird es auf die gleiche Weise wie die Spaltenwerte aggregiert.
--aggfunc: Funktion zur Aggregation. Der Standardwert ist "numpy.mean".
Betrachten Sie den folgenden Datensatz.
In [54]: import datetime
In [55]: df = pd.DataFrame({'A': ['one', 'one', 'two', 'three'] * 6,
....: 'B': ['A', 'B', 'C'] * 8,
....: 'C': ['foo', 'foo', 'foo', 'bar', 'bar', 'bar'] * 4,
....: 'D': np.random.randn(24),
....: 'E': np.random.randn(24),
....: 'F': [datetime.datetime(2013, i, 1) for i in range(1, 13)]
....: + [datetime.datetime(2013, i, 15) for i in range(1, 13)]})
....:
In [56]: df
Out[56]:
A B C D E F
0 one A foo 0.341734 -0.317441 2013-01-01
1 one B foo 0.959726 -1.236269 2013-02-01
2 two C foo -1.110336 0.896171 2013-03-01
3 three A bar -0.619976 -0.487602 2013-04-01
4 one B bar 0.149748 -0.082240 2013-05-01
.. ... .. ... ... ... ...
19 three B foo 0.690579 -2.213588 2013-08-15
20 one C foo 0.995761 1.063327 2013-09-15
21 one A bar 2.396780 1.266143 2013-10-15
22 two B bar 0.014871 0.299368 2013-11-15
23 three C bar 3.357427 -0.863838 2013-12-15
[24 rows x 6 columns]
Es ist sehr einfach, aus diesen Daten eine Pivot-Tabelle zu erstellen.
In [57]: pd.pivot_table(df, values='D', index=['A', 'B'], columns=['C'])
Out[57]:
C bar foo
A B
one A 1.120915 -0.514058
B -0.338421 0.002759
C -0.538846 0.699535
three A -1.181568 NaN
B NaN 0.433512
C 0.588783 NaN
two A NaN 1.000985
B 0.158248 NaN
C NaN 0.176180
In [58]: pd.pivot_table(df, values='D', index=['B'], columns=['A', 'C'], aggfunc=np.sum)
Out[58]:
A one three two
C bar foo bar foo bar foo
B
A 2.241830 -1.028115 -2.363137 NaN NaN 2.001971
B -0.676843 0.005518 NaN 0.867024 0.316495 NaN
C -1.077692 1.399070 1.177566 NaN NaN 0.352360
In [59]: pd.pivot_table(df, values=['D', 'E'], index=['B'], columns=['A', 'C'],
....: aggfunc=np.sum)
....:
Out[59]:
D ... E
A one three ... three two
C bar foo bar ... foo bar foo
B ...
A 2.241830 -1.028115 -2.363137 ... NaN NaN 0.128491
B -0.676843 0.005518 NaN ... -2.128743 -0.194294 NaN
C -1.077692 1.399070 1.177566 ... NaN NaN 0.872482
[3 rows x 12 columns]
Das Ergebnisobjekt ist ein "DataFrame", der wahrscheinlich einen hierarchischen Index für die Zeilen und Spalten hat. Wenn in values kein Spaltenname angegeben ist, fügt die Pivot-Tabelle der Spalte eine Hierarchieebene hinzu, die alle Daten enthält, die aggregiert werden können.
In [60]: pd.pivot_table(df, index=['A', 'B'], columns=['C'])
Out[60]:
D E
C bar foo bar foo
A B
one A 1.120915 -0.514058 1.393057 -0.021605
B -0.338421 0.002759 0.684140 -0.551692
C -0.538846 0.699535 -0.988442 0.747859
three A -1.181568 NaN 0.961289 NaN
B NaN 0.433512 NaN -1.064372
C 0.588783 NaN -0.131830 NaN
two A NaN 1.000985 NaN 0.064245
B 0.158248 NaN -0.097147 NaN
C NaN 0.176180 NaN 0.436241
Sie können auch "Grouper" für die Argumente "Index" und "Spalten" verwenden. Weitere Informationen zu "Grouper" finden Sie unter Gruppieren mit Grouper (https://pandas.pydata.org/pandas-docs/stable/user_guide/groupby.html#groupby-specify).
In [61]: pd.pivot_table(df, values='D', index=pd.Grouper(freq='M', key='F'),
....: columns='C')
....:
Out[61]:
C bar foo
F
2013-01-31 NaN -0.514058
2013-02-28 NaN 0.002759
2013-03-31 NaN 0.176180
2013-04-30 -1.181568 NaN
2013-05-31 -0.338421 NaN
2013-06-30 -0.538846 NaN
2013-07-31 NaN 1.000985
2013-08-31 NaN 0.433512
2013-09-30 NaN 0.699535
2013-10-31 1.120915 NaN
2013-11-30 0.158248 NaN
2013-12-31 0.588783 NaN
Sie können die Ausgabe einer Tabelle mit fehlenden Werten wunderschön rendern, indem Sie nach Bedarf to_string aufrufen.
In [62]: table = pd.pivot_table(df, index=['A', 'B'], columns=['C'])
In [63]: print(table.to_string(na_rep=''))
D E
C bar foo bar foo
A B
one A 1.120915 -0.514058 1.393057 -0.021605
B -0.338421 0.002759 0.684140 -0.551692
C -0.538846 0.699535 -0.988442 0.747859
three A -1.181568 0.961289
B 0.433512 -1.064372
C 0.588783 -0.131830
two A 1.000985 0.064245
B 0.158248 -0.097147
C 0.176180 0.436241
** pivot_table ist auch als Instanzmethode von DataFrame verfügbar. ** ** **
⇒ DataFrame.pivot_table()。
Zwischensumme hinzufügen
Durch Übergeben von "margin = True" an "pivot_table" wird eine spezielle Spalte und Zeile mit dem Namen "All" hinzugefügt, die die gesamte Zeilen- und Spaltenkategorie für jede Gruppe zusammenfasst.
Kreuztabelle
Mit crosstab () können Sie zwei (oder mehr) verwenden. Sie können Elemente kreuztabellieren. Wenn kein Array von Werten und Aggregatfunktionen übergeben wird, berechnet die Kreuztabelle standardmäßig die Elementhäufigkeitstabelle.
Es erhält auch die folgenden Argumente:
--index: Array etc. Werte, die nach Zeilen gruppiert werden sollen.
--columns: Array etc. Zu gruppierende Werte nach Spalte.
--Werte: Array etc. Optional. Ein Array von Werten, die entsprechend dem Element aggregiert werden sollen.
--aggfunc: Funktion. Optional. Wenn nicht angegeben, wird die Häufigkeitstabelle berechnet.
--rownames: Sequenz. Der Standardwert ist "Keine". Muss mit der Anzahl der übergebenen Zeilenarrays übereinstimmen (der Länge des im Argument index übergebenen Arrays).
--colnames: Sequenz. Der Standardwert ist "Keine". Muss mit der Anzahl der übergebenen Spaltenarrays übereinstimmen (der Länge des im Argument column übergebenen Arrays).
--margins: Boolescher Wert. Der Standardwert ist "False". Fügen Sie Zeilen und Spalten Zwischensummen hinzu.
- "Normalisieren" des booleschen Werts "{" alle "," Index "," Spalten "} ・ {0,1}. Der Standardwert ist "False". Normalisieren Sie, indem Sie alle Werte durch die Summe der Werte dividieren.
Sofern in der Kreuztabelle kein Zeilen- oder Spaltenname angegeben ist, wird das Namensattribut jeder übergebenen "Serie" verwendet.
Zum Beispiel
In [65]: foo, bar, dull, shiny, one, two = 'foo', 'bar', 'dull', 'shiny', 'one', 'two'
In [66]: a = np.array([foo, foo, bar, bar, foo, foo], dtype=object)
In [67]: b = np.array([one, one, two, one, two, one], dtype=object)
In [68]: c = np.array([dull, dull, shiny, dull, dull, shiny], dtype=object)
In [69]: pd.crosstab(a, [b, c], rownames=['a'], colnames=['b', 'c'])
Out[69]:
b one two
c dull shiny dull shiny
a
bar 1 0 0 1
foo 2 1 1 0
Wenn "Kreuztabelle" nur zwei Serien empfängt, wird eine Häufigkeitstabelle zurückgegeben.
In [70]: df = pd.DataFrame({'A': [1, 2, 2, 2, 2], 'B': [3, 3, 4, 4, 4],
....: 'C': [1, 1, np.nan, 1, 1]})
....:
In [71]: df
Out[71]:
A B C
0 1 3 1.0
1 2 3 1.0
2 2 4 NaN
3 2 4 1.0
4 2 4 1.0
In [72]: pd.crosstab(df['A'], df['B'])
Out[72]:
B 3 4
A
1 1 0
2 1 3
Wenn die übergebenen Daten kategoriale Daten enthalten, werden die ** alle ** Kategorien in die Kreuztabelle aufgenommen, auch wenn die tatsächlichen Daten keine Instanzen einer bestimmten Kategorie enthalten.
In [73]: foo = pd.Categorical(['a', 'b'], categories=['a', 'b', 'c'])
In [74]: bar = pd.Categorical(['d', 'e'], categories=['d', 'e', 'f'])
In [75]: pd.crosstab(foo, bar)
Out[75]:
col_0 d e
row_0
a 1 0
b 0 1
Normalisierung
Die Häufigkeitstabelle kann auch normalisiert werden, um Prozentsätze anstelle von Zählwerten mit dem Argument "normalisieren" anzuzeigen.
In [76]: pd.crosstab(df['A'], df['B'], normalize=True)
Out[76]:
B 3 4
A
1 0.2 0.0
2 0.2 0.6
"normalisieren" kann auch für jede Zeile oder Spalte normalisiert werden.
In [77]: pd.crosstab(df['A'], df['B'], normalize='columns')
Out[77]:
B 3 4
A
1 0.5 0.0
2 0.5 1.0
Wenn Sie die dritte "Serie" und die Aggregatfunktion ("aggfunc") an "Kreuztabelle" übergeben, wird die Funktion für den Wert in der dritten "Serie" jeder Gruppe in den ersten beiden "Serien" definiert Wird angewandt.
In [78]: pd.crosstab(df['A'], df['B'], values=df['C'], aggfunc=np.sum)
Out[78]:
B 3 4
A
1 1.0 NaN
2 1.0 2.0
Zwischensumme hinzufügen
Schließlich können Sie Zwischensummen hinzufügen und deren Ausgabe normalisieren.
In [79]: pd.crosstab(df['A'], df['B'], values=df['C'], aggfunc=np.sum, normalize=True,
....: margins=True)
....:
Out[79]:
B 3 4 All
A
1 0.25 0.0 0.25
2 0.25 0.5 0.75
All 0.50 0.5 1.00
Klasseneinteilung
Die Funktion cut () führt Gruppierungsberechnungen für Eingabearraywerte durch. Ich werde das machen. Es wird häufig verwendet, um kontinuierliche Variablen in diskrete oder kategoriale Variablen umzuwandeln.
In [80]: ages = np.array([10, 15, 13, 12, 23, 25, 28, 59, 60])
In [81]: pd.cut(ages, bins=3)
Out[81]:
[(9.95, 26.667], (9.95, 26.667], (9.95, 26.667], (9.95, 26.667], (9.95, 26.667], (9.95, 26.667], (26.667, 43.333], (43.333, 60.0], (43.333, 60.0]]
Categories (3, interval[float64]): [(9.95, 26.667] < (26.667, 43.333] < (43.333, 60.0]]
Wenn Sie eine Ganzzahl als Argument "bins" übergeben, wird ein Bin gleicher Breite gebildet. Sie können auch eine benutzerdefinierte Bin-Kante angeben.
In [82]: c = pd.cut(ages, bins=[0, 18, 35, 70])
In [83]: c
Out[83]:
[(0, 18], (0, 18], (0, 18], (0, 18], (18, 35], (18, 35], (18, 35], (35, 70], (35, 70]]
Categories (3, interval[int64]): [(0, 18] < (18, 35] < (35, 70]]
Wenn Sie einen IntervalIndex an das Argument bins übergeben, wird er zum Binieren der Daten verwendet.
pd.cut([25, 20, 50], bins=c.categories)
Berechnung von Indikatorvariablen und Dummy-Variablen
Bei Verwendung von get_dummies () werden die kategorialen Variablen "Dummy" und "Marker". Kann in "DataFrame" konvertiert werden. Beispielsweise können Sie aus einer "DataFrame" -Spalte ("Series") mit "k" verschiedenen Werten einen "DataFrame" erstellen, der "k" -Spalten enthält, die aus Einsen und Nullen bestehen.
In [84]: df = pd.DataFrame({'key': list('bbacab'), 'data1': range(6)})
In [85]: pd.get_dummies(df['key'])
Out[85]:
a b c
0 0 1 0
1 0 1 0
2 1 0 0
3 0 0 1
4 1 0 0
5 0 1 0
Das Präfixieren von Spaltennamen ist beispielsweise nützlich, wenn das Ergebnis mit dem ursprünglichen "DataFrame" zusammengeführt wird.
In [86]: dummies = pd.get_dummies(df['key'], prefix='key')
In [87]: dummies
Out[87]:
key_a key_b key_c
0 0 1 0
1 0 1 0
2 1 0 0
3 0 0 1
4 1 0 0
5 0 1 0
In [88]: df[['data1']].join(dummies)
Out[88]:
data1 key_a key_b key_c
0 0 0 1 0
1 1 0 1 0
2 2 1 0 0
3 3 0 0 1
4 4 1 0 0
5 5 0 1 0
Diese Funktion wird häufig mit diskreten Funktionen wie "Schneiden" verwendet.
In [89]: values = np.random.randn(10)
In [90]: values
Out[90]:
array([ 0.4082, -1.0481, -0.0257, -0.9884, 0.0941, 1.2627, 1.29 ,
0.0824, -0.0558, 0.5366])
In [91]: bins = [0, 0.2, 0.4, 0.6, 0.8, 1]
In [92]: pd.get_dummies(pd.cut(values, bins))
Out[92]:
(0.0, 0.2] (0.2, 0.4] (0.4, 0.6] (0.6, 0.8] (0.8, 1.0]
0 0 0 1 0 0
1 0 0 0 0 0
2 0 0 0 0 0
3 0 0 0 0 0
4 1 0 0 0 0
5 0 0 0 0 0
6 0 0 0 0 0
7 1 0 0 0 0
8 0 0 0 0 0
9 0 0 1 0 0
Siehe auch Series.str.get_dummies Bitte gib mir.
get_dummies () kann auch DataFrame empfangen. Standardmäßig werden alle kategorialen Variablen (im statistischen Sinne, dh diejenigen mit * Objekt * - oder * kategorialen * Datentypen) in Dummy-Variablen codiert.
In [93]: df = pd.DataFrame({'A': ['a', 'b', 'a'], 'B': ['c', 'c', 'b'],
....: 'C': [1, 2, 3]})
....:
In [94]: pd.get_dummies(df)
Out[94]:
C A_a A_b B_b B_c
0 1 1 0 0 1
1 2 0 1 0 1
2 3 1 0 1 0
Alle Nicht-Objektspalten werden unverändert in die Ausgabe aufgenommen. Sie können steuern, welche Spalten mit dem Argument columns codiert werden.
In [95]: pd.get_dummies(df, columns=['A'])
Out[95]:
B C A_a A_b
0 c 1 1 0
1 c 2 0 1
2 b 3 1 0
Sie können sehen, dass die Spalte "B" weiterhin in der Ausgabe enthalten ist, jedoch nicht codiert ist. Wenn Sie es nicht in die Ausgabe aufnehmen möchten, lassen Sie "B" fallen, bevor Sie "get_dummies" aufrufen.
Wie bei der "Series" -Version können Sie die Werte für "prefix" und "prefix_sep" übergeben. Standardmäßig werden Spaltennamen als Präfixe und "_" als Trennzeichen für Spaltennamen verwendet. prefix und prefix_sep können auf die folgenden drei Arten angegeben werden.
--String: Verwenden Sie in jeder zu codierenden Spalte denselben Wert für "Präfix" oder "Präfix_Sep". --List: Muss so lang sein wie die Anzahl der zu codierenden Spalten. --Dictionary: Ordnen Sie Spaltennamen Präfixen zu.
In [96]: simple = pd.get_dummies(df, prefix='new_prefix')
In [97]: simple
Out[97]:
C new_prefix_a new_prefix_b new_prefix_b new_prefix_c
0 1 1 0 0 1
1 2 0 1 0 1
2 3 1 0 1 0
In [98]: from_list = pd.get_dummies(df, prefix=['from_A', 'from_B'])
In [99]: from_list
Out[99]:
C from_A_a from_A_b from_B_b from_B_c
0 1 1 0 0 1
1 2 0 1 0 1
2 3 1 0 1 0
In [100]: from_dict = pd.get_dummies(df, prefix={'B': 'from_B', 'A': 'from_A'})
In [101]: from_dict
Out[101]:
C from_A_a from_A_b from_B_b from_B_c
0 1 1 0 0 1
1 2 0 1 0 1
2 3 1 0 1 0
Es kann nützlich sein, nur das k-1-Niveau der kategorialen Variablen beizubehalten, um eine Co-Linearität zu vermeiden, wenn die Ergebnisse in einem statistischen Modell verwendet werden. Sie können mit drop_first in diesen Modus wechseln.
In [102]: s = pd.Series(list('abcaa'))
In [103]: pd.get_dummies(s)
Out[103]:
a b c
0 1 0 0
1 0 1 0
2 0 0 1
3 1 0 0
4 1 0 0
In [104]: pd.get_dummies(s, drop_first=True)
Out[104]:
b c
0 0 0
1 1 0
2 0 1
3 0 0
4 0 0
Wenn die Spalte nur eine Ebene enthält, wird sie im Ergebnis weggelassen.
In [105]: df = pd.DataFrame({'A': list('aaaaa'), 'B': list('ababc')})
In [106]: pd.get_dummies(df)
Out[106]:
A_a B_a B_b B_c
0 1 1 0 0
1 1 0 1 0
2 1 1 0 0
3 1 0 1 0
4 1 0 0 1
In [107]: pd.get_dummies(df, drop_first=True)
Out[107]:
B_b B_c
0 0 0
1 1 0
2 0 0
3 1 0
4 0 1
Standardmäßig lautet die neue Spalte np.uint8dtype. Verwenden Sie das Argument dtype, um einen anderen Datentyp auszuwählen.
In [108]: df = pd.DataFrame({'A': list('abc'), 'B': [1.1, 2.2, 3.3]})
In [109]: pd.get_dummies(df, dtype=bool).dtypes
Out[109]:
B float64
A_a bool
A_b bool
A_c bool
dtype: object
_ Ab Version 0.23.0 _
Elementwert (Etikettencodierung)
Um einen eindimensionalen Wert als Aufzählung zu codieren, factorize () ) Verwenden Sie die.
In [110]: x = pd.Series(['A', 'A', np.nan, 'B', 3.14, np.inf])
In [111]: x
Out[111]:
0 A
1 A
2 NaN
3 B
4 3.14
5 inf
dtype: object
In [112]: labels, uniques = pd.factorize(x)
In [113]: labels
Out[113]: array([ 0, 0, -1, 1, 2, 3])
In [114]: uniques
Out[114]: Index(['A', 'B', 3.14, inf], dtype='object')
Beachten Sie, dass "factorize" "numpy.unique" ähnelt, NaN jedoch anders behandelt.
: ballot_box_with_check: ** Hinweis ** Die folgende
numpy.uniqueschlägt mitTypeErrorin Python 3 aufgrund eines Ausrichtungsfehlers fehl. Weitere Informationen finden Sie unter hier.
In [1]: x = pd.Series(['A', 'A', np.nan, 'B', 3.14, np.inf])
In [2]: pd.factorize(x, sort=True)
Out[2]:
(array([ 2, 2, -1, 3, 0, 1]),
Index([3.14, inf, 'A', 'B'], dtype='object'))
In [3]: np.unique(x, return_inverse=True)[::-1]
Out[3]: (array([3, 3, 0, 4, 1, 2]), array([nan, 3.14, inf, 'A', 'B'], dtype=object))
: ballot_box_with_check: ** Hinweis ** Wenn Sie eine Spalte als kategoriale Variable (wie den R-Faktor) behandeln möchten, ist
df [" cat_col "] = pd.Categorical (df [" col "])oderdf ["cat_col"] = Sie können df ["col"] .astype ("category")verwenden. Eine vollständige Dokumentation zuCategoricalfinden Sie unter Einführung in Categorical. //pandas.pydata.org/pandas-docs/stable/user_guide/categorical.html#categorical) und API-Dokumentation Siehe # api-arrays-kategorisch).
Beispiel
Dieser Abschnitt enthält häufig gestellte Fragen und Beispiele. Die Spaltennamen und zugehörigen Spaltenwerte haben Namen, die der Drehung dieses DataFrame in den folgenden Antworten entsprechen.
In [115]: np.random.seed([3, 1415])
In [116]: n = 20
In [117]: cols = np.array(['key', 'row', 'item', 'col'])
In [118]: df = cols + pd.DataFrame((np.random.randint(5, size=(n, 4))
.....: // [2, 1, 2, 1]).astype(str))
.....:
In [119]: df.columns = cols
In [120]: df = df.join(pd.DataFrame(np.random.rand(n, 2).round(2)).add_prefix('val'))
In [121]: df
Out[121]:
key row item col val0 val1
0 key0 row3 item1 col3 0.81 0.04
1 key1 row2 item1 col2 0.44 0.07
2 key1 row0 item1 col0 0.77 0.01
3 key0 row4 item0 col2 0.15 0.59
4 key1 row0 item2 col1 0.81 0.64
.. ... ... ... ... ... ...
15 key0 row3 item1 col1 0.31 0.23
16 key0 row0 item2 col3 0.86 0.01
17 key0 row4 item0 col3 0.64 0.21
18 key2 row2 item2 col0 0.13 0.45
19 key0 row2 item0 col4 0.37 0.70
[20 rows x 6 columns]
Pivot mit einem einzigen Aggregat
Angenommen, Sie möchten "df" so drehen, dass der Wert von "col" die Spalte ist, der Wert von "row" der Index ist und der Durchschnitt von "val0" der Tabellenwert ist. Zu diesem Zeitpunkt sieht der resultierende DataFrame folgendermaßen aus:
col col0 col1 col2 col3 col4
row
row0 0.77 0.605 NaN 0.860 0.65
row2 0.13 NaN 0.395 0.500 0.25
row3 NaN 0.310 NaN 0.545 NaN
row4 NaN 0.100 0.395 0.760 0.24
Verwenden Sie dazu pivot_table (). Beachten Sie, dass "aggfunc =" mean "das Standardverhalten ist, obwohl hier ausdrücklich erwähnt.
In [122]: df.pivot_table(
.....: values='val0', index='row', columns='col', aggfunc='mean')
.....:
Out[122]:
col col0 col1 col2 col3 col4
row
row0 0.77 0.605 NaN 0.860 0.65
row2 0.13 NaN 0.395 0.500 0.25
row3 NaN 0.310 NaN 0.545 NaN
row4 NaN 0.100 0.395 0.760 0.24
Sie können auch den Parameter fill_value verwenden, um fehlende Werte zu ersetzen.
In [123]: df.pivot_table(
.....: values='val0', index='row', columns='col', aggfunc='mean', fill_value=0)
.....:
Out[123]:
col col0 col1 col2 col3 col4
row
row0 0.77 0.605 0.000 0.860 0.65
row2 0.13 0.000 0.395 0.500 0.25
row3 0.00 0.310 0.000 0.545 0.00
row4 0.00 0.100 0.395 0.760 0.24
Beachten Sie auch, dass Sie andere Aggregatfunktionen übergeben können. Zum Beispiel können Sie "sum" übergeben.
In [124]: df.pivot_table(
.....: values='val0', index='row', columns='col', aggfunc='sum', fill_value=0)
.....:
Out[124]:
col col0 col1 col2 col3 col4
row
row0 0.77 1.21 0.00 0.86 0.65
row2 0.13 0.00 0.79 0.50 0.50
row3 0.00 0.31 0.00 1.09 0.00
row4 0.00 0.10 0.79 1.52 0.24
Eine andere Aggregation besteht darin, zu berechnen, wie oft Spalten und Zeilen gleichzeitig auftreten (als "Kreuzaggregation" bezeichnet). Übergeben Sie dazu size an den Parameter aggfunc.
In [125]: df.pivot_table(index='row', columns='col', fill_value=0, aggfunc='size')
Out[125]:
col col0 col1 col2 col3 col4
row
row0 1 2 0 1 1
row2 1 0 2 1 2
row3 0 1 0 2 0
row4 0 1 2 2 1
Pivot mit mehreren Aggregaten
Sie können auch mehrere Aggregationen durchführen. Sie können beispielsweise eine Liste an das Argument "aggfunc" übergeben, um sowohl die Gesamtsumme als auch den Durchschnittswert auszuführen.
In [126]: df.pivot_table(
.....: values='val0', index='row', columns='col', aggfunc=['mean', 'sum'])
.....:
Out[126]:
mean sum
col col0 col1 col2 col3 col4 col0 col1 col2 col3 col4
row
row0 0.77 0.605 NaN 0.860 0.65 0.77 1.21 NaN 0.86 0.65
row2 0.13 NaN 0.395 0.500 0.25 0.13 NaN 0.79 0.50 0.50
row3 NaN 0.310 NaN 0.545 NaN NaN 0.31 NaN 1.09 NaN
row4 NaN 0.100 0.395 0.760 0.24 NaN 0.10 0.79 1.52 0.24
Sie können eine Liste an den Parameter values übergeben, um sie über mehrere Wertespalten zu aggregieren.
In [127]: df.pivot_table(
.....: values=['val0', 'val1'], index='row', columns='col', aggfunc=['mean'])
.....:
Out[127]:
mean
val0 val1
col col0 col1 col2 col3 col4 col0 col1 col2 col3 col4
row
row0 0.77 0.605 NaN 0.860 0.65 0.01 0.745 NaN 0.010 0.02
row2 0.13 NaN 0.395 0.500 0.25 0.45 NaN 0.34 0.440 0.79
row3 NaN 0.310 NaN 0.545 NaN NaN 0.230 NaN 0.075 NaN
row4 NaN 0.100 0.395 0.760 0.24 NaN 0.070 0.42 0.300 0.46
Sie können eine Liste als Spaltenparameter übergeben, um sie in mehrere Spalten zu unterteilen.
In [128]: df.pivot_table(
.....: values=['val0'], index='row', columns=['item', 'col'], aggfunc=['mean'])
.....:
Out[128]:
mean
val0
item item0 item1 item2
col col2 col3 col4 col0 col1 col2 col3 col4 col0 col1 col3 col4
row
row0 NaN NaN NaN 0.77 NaN NaN NaN NaN NaN 0.605 0.86 0.65
row2 0.35 NaN 0.37 NaN NaN 0.44 NaN NaN 0.13 NaN 0.50 0.13
row3 NaN NaN NaN NaN 0.31 NaN 0.81 NaN NaN NaN 0.28 NaN
row4 0.15 0.64 NaN NaN 0.10 0.64 0.88 0.24 NaN NaN NaN NaN
Erweitern Sie die Spalten der Liste
_ Ab Version 0.25.0 _
Die Spaltenwerte können wie eine Liste aussehen.
In [129]: keys = ['panda1', 'panda2', 'panda3']
In [130]: values = [['eats', 'shoots'], ['shoots', 'leaves'], ['eats', 'leaves']]
In [131]: df = pd.DataFrame({'keys': keys, 'values': values})
In [132]: df
Out[132]:
keys values
0 panda1 [eats, shoots]
1 panda2 [shoots, leaves]
2 panda3 [eats, leaves]
Verwenden Sie explode () für values Sie können Spalten "explodieren" und jedes Listenelement in eine separate Zeile konvertieren. Dadurch wird der Indexwert aus der ursprünglichen Zeile dupliziert.
In [133]: df['values'].explode()
Out[133]:
0 eats
0 shoots
1 shoots
1 leaves
2 eats
2 leaves
Name: values, dtype: object
Sie können auch die Spalten von "DataFrame" erweitern.
In [134]: df.explode('values')
Out[134]:
keys values
0 panda1 eats
0 panda1 shoots
1 panda2 shoots
1 panda2 leaves
2 panda3 eats
2 panda3 leaves
Series.explode () ist eine leere Liste Ersetzen Sie durch "np.nan" und behalten Sie den skalaren Eintrag bei. Der resultierende Datentyp "Serie" ist immer "Objekt".
In [135]: s = pd.Series([[1, 2, 3], 'foo', [], ['a', 'b']])
In [136]: s
Out[136]:
0 [1, 2, 3]
1 foo
2 []
3 [a, b]
dtype: object
In [137]: s.explode()
Out[137]:
0 1
0 2
0 3
1 foo
2 NaN
3 a
3 b
dtype: object
Angenommen, eine Spalte hat eine durch Kommas getrennte Zeichenfolge und Sie möchten sie erweitern.
In [138]: df = pd.DataFrame([{'var1': 'a,b,c', 'var2': 1},
.....: {'var1': 'd,e,f', 'var2': 2}])
.....:
In [139]: df
Out[139]:
var1 var2
0 a,b,c 1
1 d,e,f 2
Sie können problemlos einen vertikalen DataFrame erstellen, indem Sie Erweiterungs- und Kettenoperationen ausführen.
In [140]: df.assign(var1=df.var1.str.split(',')).explode('var1')
Out[140]:
var1 var2
0 a 1
0 b 1
0 c 1
1 d 2
1 e 2
1 f 2