Apprendre Python avec ChemTHEATER 02
Partie 2 Estimation statistique
Chap.0 Débit global
La partie 2 fait une estimation statistique. En sciences naturelles, il n'est pas possible d'enquêter directement sur tous les objets à étudier (la nature), donc l'investigation d'échantillons est courante.
À ce moment, l'estimation statistique consiste à estimer la distribution de l'ensemble de la population inconnue à partir de l'échantillon obtenu (une partie de la population).
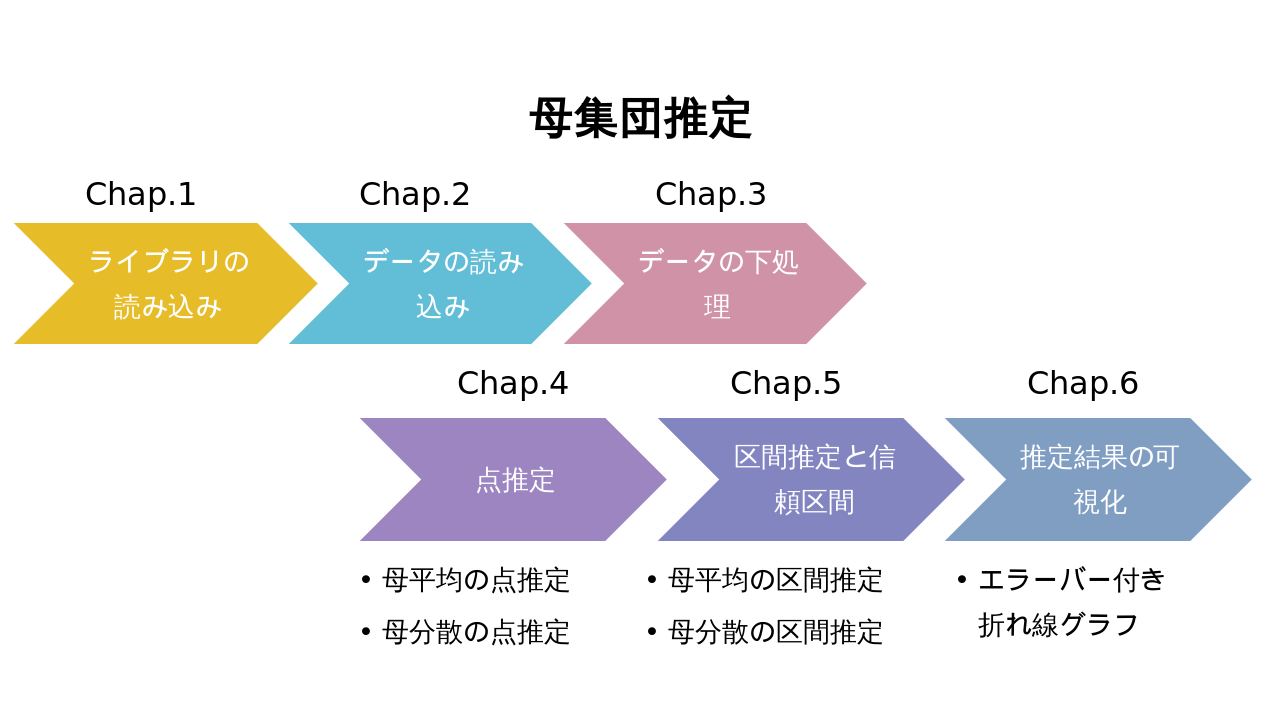
Chargement de la bibliothèque Chap.1
%matplotlib inline
import numpy as np
from scipy import stats
import math
import pandas as pd
from matplotlib import pyplot as plt
Commencez par charger les bibliothèques requises. La première ligne est une commande magique pour afficher matplotlib dans Jupyter Notebook comme dans la partie 1.
La deuxième ligne et les suivantes sont les bibliothèques utilisées cette fois. Parmi ces bibliothèques, la bibliothèque mathématique est intégrée à Python en standard. D'autres bibliothèques sont installées dans Anaconda.
Soit dit en passant, les mathématiques et Numpy ont des fonctions similaires, mais les mathématiques sont une fonction intégrée à Python standard, et Numpy est un module d'extension qui rend les calculs numériques complexes efficaces, et leurs rôles sont différents.
| Bibliothèque | Présentation | But de l'utilisation cette fois | URL officielle |
|---|---|---|---|
| NumPy | Bibliothèque de calculs numériques | Utilisé pour le calcul numérique dans le traitement statistique | https://www.numpy.org |
| Scipy | Bibliothèque de calculs scientifiques | Utilisé pour calculer des estimations statistiques | https://www.scipy.org |
| math | Bibliothèque de calcul numérique standard | Utilisé pour des calculs simples tels que les racines carrées | https://docs.python.org/ja/3/library/math.html |
| pandas | Bibliothèque d'analyse des données | Utilisé pour la lecture et le formatage des données | https://pandas.pydata.org |
| Matplotlib | Bibliothèque de dessins graphiques | Utilisé pour la visualisation des données | https://matplotlib.org |
Lecture des données Chap.2
Cette fois, nous utiliserons les données de Katsuo ( Katsuwonus pelamis ). Reportez-vous au chapitre 4 de la partie 0 et téléchargez les données de mesure des données d'échantillon de bonite à partir de la recherche d'échantillons de ChemTHEATER.
Une fois téléchargés, déplacez les données mesurées et les données d'échantillons dans le dossier contenant ce fichier notebook. Après cela, après avoir redémarré Anaconda, lisez à la fois les données de mesure et les exemples de données à l'aide de la fonction read_csv des pandas comme dans la partie 1.
data_file = "measureddata_20190930045953.tsv" #Remplacez la chaîne de caractères saisie dans la variable par le nom de fichier tsv de vos données mesurées
chem = pd.read_csv(data_file, delimiter="\t")
chem = chem.drop(["ProjectID", "ScientificName", "RegisterDate", "UpdateDate"], axis=1) #Supprimer les colonnes en double lors de la jonction avec des échantillons ultérieurement
sample_file = "samples_20190930045950.tsv" #Remplacez la chaîne de caractères entrée dans la variable par le nom de fichier tsv de vos échantillons
sample = pd.read_csv(sample_file, delimiter="\t")
Lors de la lecture ou du traitement d'un fichier par programmation comme python, il est préférable de prendre l'habitude de vérifier si le fichier peut être lu comme prévu. En passant, dans le cas de Jupyter Notebook, si vous entrez uniquement le nom de la variable, le contenu de cette variable sera affiché dans Out, ce qui est pratique.
chem
| MeasuredID | SampleID | ChemicalID | ChemicalName | ExperimentID | MeasuredValue | AlternativeData | Unit | Remarks | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | SAA000001 | CH0000096 | ΣPCBs | EXA000001 | 6.659795 | NaN | ng/g wet | NaN |
| 1 | 2 | SAA000002 | CH0000096 | ΣPCBs | EXA000001 | 9.778107 | NaN | ng/g wet | NaN |
| 2 | 3 | SAA000003 | CH0000096 | ΣPCBs | EXA000001 | 5.494933 | NaN | ng/g wet | NaN |
| 3 | 4 | SAA000004 | CH0000096 | ΣPCBs | EXA000001 | 7.354636 | NaN | ng/g wet | NaN |
| 4 | 5 | SAA000005 | CH0000096 | ΣPCBs | EXA000001 | 9.390950 | NaN | ng/g wet | NaN |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 74 | 75 | SAA000082 | CH0000096 | ΣPCBs | EXA000001 | 3.321208 | NaN | ng/g wet | NaN |
| 75 | 76 | SAA000083 | CH0000096 | ΣPCBs | EXA000001 | 3.285111 | NaN | ng/g wet | NaN |
| 76 | 77 | SAA000084 | CH0000096 | ΣPCBs | EXA000001 | 0.454249 | NaN | ng/g wet | NaN |
| 77 | 78 | SAA000085 | CH0000096 | ΣPCBs | EXA000001 | 0.100000 | <1.00E-1 | ng/g wet | NaN |
| 78 | 79 | SAA000086 | CH0000096 | ΣPCBs | EXA000001 | 0.702224 | NaN | ng/g wet | NaN |
79 rows × 9 columns
sample
| ProjectID | SampleID | SampleType | TaxonomyID | UniqCodeType | UniqCode | SampleName | ScientificName | CommonName | CollectionYear | ... | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | PRA000001 | SAA000001 | ST008 | 8226 | es-BANK | EF00564 | NaN | Katsuwonus pelamis | Skipjack tuna | 1998 | ... |
| 1 | PRA000001 | SAA000002 | ST008 | 8226 | es-BANK | EF00565 | NaN | Katsuwonus pelamis | Skipjack tuna | 1998 | ... |
| 2 | PRA000001 | SAA000003 | ST008 | 8226 | es-BANK | EF00566 | NaN | Katsuwonus pelamis | Skipjack tuna | 1998 | ... |
| 3 | PRA000001 | SAA000004 | ST008 | 8226 | es-BANK | EF00567 | NaN | Katsuwonus pelamis | Skipjack tuna | 1998 | ... |
| 4 | PRA000001 | SAA000005 | ST008 | 8226 | es-BANK | EF00568 | NaN | Katsuwonus pelamis | Skipjack tuna | 1998 | ... |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 74 | PRA000001 | SAA000082 | ST008 | 8226 | es-BANK | EF00616 | NaN | Katsuwonus pelamis | Skipjack tuna | 1999 | ... |
| 75 | PRA000001 | SAA000083 | ST008 | 8226 | es-BANK | EF00617 | NaN | Katsuwonus pelamis | Skipjack tuna | 1999 | ... |
| 76 | PRA000001 | SAA000084 | ST008 | 8226 | es-BANK | EF00619 | NaN | Katsuwonus pelamis | Skipjack tuna | 1999 | ... |
| 77 | PRA000001 | SAA000085 | ST008 | 8226 | es-BANK | EF00620 | NaN | Katsuwonus pelamis | Skipjack tuna | 1999 | ... |
| 78 | PRA000001 | SAA000086 | ST008 | 8226 | es-BANK | EF00621 | NaN | Katsuwonus pelamis | Skipjack tuna | 1999 | ... |
79 rows × 66 columns
Chap.3 Préparation des données
Après avoir lu les données, l'étape suivante consiste à préparer les données.
Tout d'abord, les données (chim et échantillon) divisées en deux sont intégrées, et seules les données nécessaires sont extraites. Cette fois, je veux utiliser les données du ΣPCB de Katsuo, donc je vais extraire uniquement les données dont la valeur dans la colonne "ChemicalName" est "ΣPCB".
df = pd.merge(chem, sample, on="SampleID")
data = df[df["ChemicalName"] == "ΣPCBs"]
Ensuite, vérifiez si l'unité des données de mesure est différente. En effet, si les unités de données sont différentes comme dans la partie 1, il n'est pas possible de simplement comparer et intégrer.
data["Unit"].unique()
array(['ng/g wet'], dtype=object)
Vous pouvez utiliser la méthode unique
pandas pour voir une liste des valeurs contenues dans ce bloc de données. Ici, si vous sortez une liste de valeurs incluses dans la colonne "Unit", vous pouvez voir qu'il s'agit uniquement de "ng / g wet", donc cette fois il n'est pas nécessaire de diviser les données par unité. </ p>
Enfin, la colonne avec seulement N / A est supprimée et la préparation des données est terminée.
data = data.dropna(how='all', axis=1)
data
| MeasuredID | SampleID | ChemicalID | ChemicalName | ExperimentID | MeasuredValue | AlternativeData | Unit | ProjectID | SampleType | ... | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | SAA000001 | CH0000096 | ΣPCBs | EXA000001 | 6.659795 | NaN | ng/g wet | PRA000001 | ST008 | ... |
| 1 | 2 | SAA000002 | CH0000096 | ΣPCBs | EXA000001 | 9.778107 | NaN | ng/g wet | PRA000001 | ST008 | ... |
| 2 | 3 | SAA000003 | CH0000096 | ΣPCBs | EXA000001 | 5.494933 | NaN | ng/g wet | PRA000001 | ST008 | ... |
| 3 | 4 | SAA000004 | CH0000096 | ΣPCBs | EXA000001 | 7.354636 | NaN | ng/g wet | PRA000001 | ST008 | ... |
| 4 | 5 | SAA000005 | CH0000096 | ΣPCBs | EXA000001 | 9.390950 | NaN | ng/g wet | PRA000001 | ST008 | ... |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 74 | 75 | SAA000082 | CH0000096 | ΣPCBs | EXA000001 | 3.321208 | NaN | ng/g wet | PRA000001 | ST008 | ... |
| 75 | 76 | SAA000083 | CH0000096 | ΣPCBs | EXA000001 | 3.285111 | NaN | ng/g wet | PRA000001 | ST008 | ... |
| 76 | 77 | SAA000084 | CH0000096 | ΣPCBs | EXA000001 | 0.454249 | NaN | ng/g wet | PRA000001 | ST008 | ... |
| 77 | 78 | SAA000085 | CH0000096 | ΣPCBs | EXA000001 | 0.100000 | <1.00E-1 | ng/g wet | PRA000001 | ST008 | ... |
| 78 | 79 | SAA000086 | CH0000096 | ΣPCBs | EXA000001 | 0.702224 | NaN | ng/g wet | PRA000001 | ST008 | ... |
79 rows × 35 columns
Estimation en points Chap.4
Parmi les estimations statistiques qui estiment la population à partir de l'échantillon, l'estimation ponctuelle est l'estimation précise de la valeur. Ici, la concentration en ΣPCB de la population (individu entier dans la zone de collecte) est estimée à partir de l'échantillon de la concentration en ΣPCB détectée dans la bonite.
Tout d'abord, vérifiez combien d'années de données sont incluses afin de calculer chaque année et voyez la transition du changement.
data['CollectionYear'].unique()
array([1998, 1997, 1999, 2001], dtype=int64)
À partir de la méthode unique ci-dessus, il a été constaté que l'ensemble de données contient des données pour les trois années 1997-1999 et 2001. Prenons d'abord les données de 1997. À ce stade, modifiez les données extraites au format ndarray 1 de Numpy afin que les calculs futurs soient plus faciles.
De même, les données de 1998 et 1999 sont également extraites.
Ici, les estimations non biaisées de la moyenne et de la variance sont calculées. De même, calculez l'estimation sans biais de la variance. Notez que la fonction var de Numpy peut calculer les deux variances et que la variance non biaisée est sortie avec le paramètre ddof = 1. Cependant, notez que la distribution d'échantillon de ddof = 0 est sortie par défaut. De même, calculez les estimations sans biais de la moyenne et de la variance pour 1998 et 1999.
Ici, nous résumerons les valeurs représentatives obtenues. Premièrement,
Au Chap.5, contrairement à l'estimation ponctuelle obtenue au Chap.4, l'estimation d'intervalle est effectuée pour estimer la moyenne de la population et la variance de la population dans une fourchette statistiquement constante.
Tout d'abord, vérifiez le nombre de données dans l'ensemble de données de chaque année avant d'estimer l'intervalle. Le nombre de données peut être calculé en utilisant la fonction len implémentée en standard en python.
De ce qui précède, le nombre total de données dans l'ensemble de données pour chaque année de 1997 à 1999 est connu.
Parmi ceux-ci, l'ensemble de données de 1997 est un grand échantillon de $ n = 50 $, et les ensembles de données de 1998 et 1999 sont respectivement $ n = \ left \ {\ begin {array} {ll}. 15 & \ left (1998 \ right) \\ 13 & \ left (1999 \ right) \ end {array} \ right. $, Qui est un petit échantillon. Premièrement, l'intervalle de la moyenne de la population est estimé à partir de l'ensemble de données de 1997. Dans ce cas, comme la variance de la population est inconnue et un grand échantillon ($ n> 30 $), la moyenne de l'échantillon ($ \ override X $) est une distribution normale $ N \ left (\ mu, \ frac {s ^ 2} { n} \ right) $ peut être approximé. Par conséquent, si la moyenne de la population est estimée par la fiabilité ($ \ alpha $), l'intervalle de confiance sera comme indiqué dans l'équation suivante.
$\overline X - z_\frac{\alpha}{2} \sqrt{\frac{s^2}{n}} < \mu < \overline X - z_\frac{\alpha}{2} \sqrt{\frac{s^2}{n}} $
En python, si vous utilisez stas.norm.interval () de Scipy, vous pouvez obtenir la plage de alpha × 100% dans la distribution normale de la moyenne (loc) et de l'écart type (échelle) centrée sur la valeur médiane. Ensuite, nous estimons l'intervalle moyen de population pour les ensembles de données de 1998 et 1999. Ce sont de petits échantillons ($ n \ leq 30 $) avec une variance de population inconnue. Dans ce cas, la moyenne de population $ \ mu $ n'est pas la distribution normale $ N \ left (\ mu, \ frac {s ^ 2} {n} \ right) $, mais la distribution t avec degrés de liberté ($ n-1 $). Utiliser. Par conséquent, si la moyenne de la population est estimée par la fiabilité ($ \ alpha $), l'intervalle de confiance sera comme indiqué dans l'équation suivante.
$\overline X - t_\frac{\alpha}{2}\left(n-1\right)\sqrt{\frac{s^2}{n}} < \mu < \overline X + t_\frac{\alpha}{2}\left(n-1\right)\sqrt{\frac{s^2}{n}} $
En python, si vous utilisez stats.t.interval (), la plage où la distribution t de la moyenne (loc), de l'écart type (échelle) et du degré de liberté (df) est alpha × 100% est centrée sur la valeur médiane. Peut être obtenu comme. L'intervalle de confiance de 95% signifie que la moyenne de la population se situe dans cette plage avec une probabilité de 95%. Autrement dit, si la fiabilité ($ \ alpha $) est réduite, la probabilité que la moyenne de la population soit incluse dans l'intervalle de confiance est réduite, et en même temps, l'intervalle de confiance est réduit.
Ensuite, l'estimation d'intervalle de la variance de la population est effectuée. Dans l'estimation par intervalles de la variance de la population ($ \ sigma ^ 2 $), $ \ frac {\ left (n-1 \ right) s ^ 2} {\ sigma ^ 2} $ a un degré de liberté $ (n-1) $. Utilisez pour suivre la distribution $ \ chi ^ 2 $ de.
$\chi_\frac{\alpha}{2}\left(n-1\right) \leq \frac{\left( n-1 \right)s^2}{\sigma^2} \leq \chi_{1-\frac{\alpha}{2}}\left(n-1\right)$
Premièrement, le point de pourcentage ($ \ chi_ \ frac {\ alpha} {2} \ left (n-1 \ right), \ chi_ de la distribution $ \ chi ^ 2 $ de la liberté ($ n-1 $) Recherchez {1- \ frac {\ alpha} {2}} \ left (n-1 \ right) $). Ici, la fiabilité est de 0,95. Ensuite, trouvez l'intervalle de confiance. Reportez-vous à la formule suivante pour la dérivation.
$\frac{\left(n-1\right)s^2}{\chi_\frac{\alpha}{2}\left(n-1\right)} \leq \sigma^2 \leq \frac{\left(n-1\right)s^2}{\chi_{1-\frac{\alpha}{2}}\left(n-1\right)}$
De même, pour les ensembles de données de 1998 et 1999, l'estimation d'intervalle d'intervalle de la variance est effectuée. De plus, contrairement à l'estimation par intervalles de la moyenne de la population, la distribution de $ \ frac {\ left (n-1 \ right) s ^ 2} {\ sigma ^ 2} $ est $ \ chi ^ 2 quelle que soit la taille de l'échantillon. Suivez la distribution $.
Maintenant, visualisons la moyenne de la population estimée aux Chap.4 et Chap.5 sur un graphique.
Premièrement, les valeurs de l'estimation ponctuelle moyenne de la population au chapitre 4 sont résumées en séries chronologiques.
Ensuite, l'intervalle de confiance de la moyenne de la population avec une fiabilité de 95% estimée au chapitre 5 est également résumé en séries chronologiques.
L'intervalle de confiance à 95% de la moyenne de la population ne peut pas être utilisé pour la visualisation tel quel, donc la largeur de l'intervalle de confiance est calculée.
Enfin, visualisez avec matplotlib. L'intervalle de confiance est affiché avec une barre d'erreur. Lors de l'affichage de la barre d'erreur avec matplotlib, utilisez la méthode errorbar. 1 Un format de données qui stocke la matrice d'ordre n dans Numpy.
Recommended Posts
pcb_1997 = np.array(data[data['CollectionYear']==1997]["MeasuredValue"]) #Extraire uniquement les mesures de 1997
pcb_1997
array([ 10.72603788, 9.22208078, 7.59790835, 30.95079465,
15.27462553, 14.15719633, 13.28955903, 14.87712806,
9.86650189, 18.26554514, 3.39951845, 6.58172781,
12.43564814, 6.1948639 , 6.41605666, 4.98827291,
12.36669815, 31.17955551, 8.16184346, 4.60893266,
36.85826409, 52.99841724, 39.22500351, 53.92302702,
69.4308048 , 73.97686479, 125.3887794 , 45.39974771,
54.12726127, 39.77794045, 101.2736126 , 38.06220403,
126.8301693 , 70.25308435, 31.24246301, 21.3958656 ,
41.85726522, 30.91112132, 81.12597135, 10.76755148,
24.20442213, 24.57497594, 14.84353549, 59.53687389,
52.78443082, 8.4644697 , 4.15293758, 3.31957452,
4.51832675, 6.98373973])
pcb_1998 = np.array(data[data['CollectionYear']==1998]["MeasuredValue"]) #Extraire seulement les mesures de 1998
pcb_1999 = np.array(data[data['CollectionYear']==1999]["MeasuredValue"]) #Extraire seulement 1999 mesures
Le premier est l'estimation sans biais de la moyenne ($ \ hat {\ mu} $), qui tire parti du fait que la valeur attendue de la moyenne de l'échantillon ($ \ override {X} $) est égale à la moyenne de la population. (Voir la formule ci-dessous)
$$ E \left(\overline X \right) = E\left(\frac{1}{n} \sum_{i=1}^{n} \left(x_i\right)\right) = \frac{1}{n}\sum_{i=1}^{n} E\left(x_i\right) = \frac{1}{n} \times n\mu = \mu \\ \therefore \hat\mu = \overline{x} $$s_mean_1997 = np.mean(pcb_1997)
s_mean_1997
31.775384007760003
À ce stade, la valeur attendue de la variance de l'échantillon ($ S ^ 2 $) ne prend pas la même valeur que la variance de la population ($ \ sigma ^ 2 $), et il est nécessaire d'obtenir la variance sans biais ($ s ^ 2 $) à la place. Faire attention à.
$$\hat\sigma^2 \neq S^2 = \frac{1}{n} \sum_{i=1}^{n} \left( x_i - \overline X \right) \\ \hat\sigma^2 = s^2 = \frac{1}{n-1} \sum_{i=1}^{n} \left( x_i - \overline X \right)$$
$$\mathrm{np.var}\left(x_1 \ldots x_n, \mathrm{ddof=0}\right): S^2 = \frac{1}{n} \sum_{i=1}^{n} \left( x_i - \overline X \right) \\
\mathrm{np.var}\left(x_1 \ldots x_n, \mathrm{ddof=1}\right): \hat\sigma^2 = \frac{1}{n-1} \sum_{i=1}^{n} \left( x_i - \overline X \right)$$u_var_1997 = np.var(pcb_1997, ddof=1)
u_var_1997
942.8421749786518
s_mean_1998, s_mean_1999 = np.mean(pcb_1998), np.mean(pcb_1999)
u_var_1998, u_var_1999 = np.var(pcb_1998, ddof=1), np.var(pcb_1999, ddof=1)
s_mean_1997, s_mean_1998, s_mean_1999
(31.775384007760003, 17.493267312533337, 30.583242522000003)
u_var_1997, u_var_1998, u_var_1999
(942.8421749786518, 240.2211176248311, 1386.7753819003349)
Chapitre 5 Section d'estimation et de confiance
Sec.5-1 Estimation d'intervalle de la moyenne de la population
n_1997 = len(pcb_1997)
n_1997
50
n_1998, n_1999 = len(pcb_1998), len(pcb_1999)
n_1998, n_1999
(15, 13)
Par conséquent, il convient de noter que le traitement de l'estimation d'intervalle après cela est légèrement différent.
Ici, l'intervalle de confiance est calculé par la fiabilité ($ \ alpha = 0,95 $).
m_interval_1997 = stats.norm.interval(alpha=0.95, loc=s_mean_1997, scale=math.sqrt(pcb_1997.var(ddof=1)/n_1997))
m_interval_1997
(23.26434483549182, 40.28642318002819)
Ici, l'intervalle de confiance est calculé en fonction de la fiabilité ($ \ alpha = 0,95 $).
m_interval_1998 = stats.t.interval(alpha=0.95, df=n_1998-1, loc=s_mean_1998, scale=math.sqrt(pcb_1998.var(ddof=1)/n_1998))
m_interval_1999 = stats.t.interval(alpha=0.95, df=n_1999-1, loc=s_mean_1999, scale=math.sqrt(pcb_1999.var(ddof=1)/n_1999))
m_interval_1997, m_interval_1998, m_interval_1999
((23.26434483549182, 40.28642318002819),
(8.910169386248537, 26.076365238818138),
(8.079678286109523, 53.086806757890486))
stats.norm.interval(alpha=0.9, loc=s_mean_1997, scale=math.sqrt(pcb_1997.var(ddof=1)/n_1997))
(24.63269477364296, 38.91807324187704)
Section 5-2 Estimation de la variance de la population
En python, vous pouvez obtenir la plage de alpha x 100% du degré de liberté (df) avec stats.chi2.interval () de Scipy.
chi_025_1997, chi_975_1997 = stats.chi2.interval(alpha=0.95, df=n_1997-1)
chi_025_1997, chi_975_1997
(31.554916462667137, 70.22241356643451)
v_interval_1997 = (n_1997 - 1)*np.var(pcb_1997, ddof=1) / chi_975_1997, (n_1997 - 1)*np.var(pcb_1997, ddof=1) / chi_025_1997
v_interval_1997
(657.8991553778869, 1464.0909168183869)
chi_025_1998, chi_975_1998 = stats.chi2.interval(alpha=0.95, df=n_1998-1)
chi_025_1999, chi_975_1999 = stats.chi2.interval(alpha=0.95, df=n_1999-1)
v_interval_1998 = (n_1998 - 1)*np.var(pcb_1998, ddof=1) / chi_975_1998, (n_1998 - 1)*np.var(pcb_1998, ddof=1) / chi_025_1998
v_interval_1999 = (n_1999 - 1)*np.var(pcb_1999, ddof=1) / chi_975_1999, (n_1999 - 1)*np.var(pcb_1999, ddof=1) / chi_025_1999
v_interval_1997, v_interval_1998, v_interval_1999
((657.8991553778869, 1464.0909168183869),
(128.76076176378118, 597.4878836139195),
(713.0969734866349, 3778.8609867211235))
chi_025_1997, chi_975_1997 = stats.chi2.interval(alpha=0.9, df=n_1997-1)
(n_1997 - 1)*np.var(pcb_1997, ddof=1) / chi_975_1997, (n_1997 - 1)*np.var(pcb_1997, ddof=1) / chi_025_1997
(696.4155490924242, 1361.5929987004467)
Chap.6 Visualisation des résultats d'estimation
x_list = [1997, 1998, 1999]
y_list = [s_mean_1997, s_mean_1998, s_mean_1999]
interval_list = []
interval_list.append(m_interval_1997)
interval_list.append(m_interval_1998)
interval_list.append(m_interval_1999)
interval_list
[(23.26434483549182, 40.28642318002819),
(8.910169386248537, 26.076365238818138),
(8.079678286109523, 53.086806757890486)]
interval_list = np.array(interval_list).T[1] - y_list
x_list, y_list, interval_list
([1997, 1998, 1999],
[31.775384007760003, 17.493267312533337, 30.583242522000003],
array([ 8.51103917, 8.58309793, 22.50356424]))
Dans cette méthode, la valeur de l'axe X (ici, l'année), la valeur de l'axe Y (ici, la moyenne de la population de l'estimation ponctuelle) et la longueur de la barre d'erreur (ici, la largeur de l'intervalle de confiance) sont spécifiées.
fig = plt.figure()
ax = fig.add_subplot(1,1,1)
ax.errorbar(x=x_list, y=y_list, yerr=interval_list, fmt='o-', capsize=4, ecolor='red')
plt.xticks(x_list)
ax.set_title("Katsuwonus pelamis")
ax.set_ylabel("ΣPCBs [ng/g wet]")
plt.show()

note de bas de page