[PYTHON] Ich habe versucht zu überprüfen, wie schnell der Mnist des Chainer-Beispiels mit Cython beschleunigt werden kann
Warum machen
Es wurde seit Chainer 1.5 beschleunigt. Es wurde erwähnt, dass die Verwendung von Cython einer der Gründe für die Beschleunigung war, daher hatte ich eine einfache Frage, wie schnell es sein würde, wenn ich das Beispiel mit Cython umschreiben würde, also habe ich es versucht.
PC-Spezifikationen
OS:OS X Yosemite CPU: 2.7GHz Intel Core i5 Memory:8GHz DDR3
Bedingungen
Verwenden Sie Beispiel Mnist Anzahl der Lernvorgänge: 20 Mal Die Daten wurden im Voraus heruntergeladen
Visualisierung
Visualisierung mit Profiler
Verwenden Sie Pycallgraph
http://pycallgraph.slowchop.com
Installieren Sie graphviz
http://www.graphviz.org/Download_macos.php
Installieren Sie X11 (für Yosemite)
http://www.xquartz.org/
Wenn fehlgeschlagen mit Fehlercode 256 erscheint
https://github.com/gak/pycallgraph/issues/100
Bei Verwendung von Pycallgraph
python pycallgraph graphviz -- ./Dateiname.py
Was ich machen will; was ich vorhabe zu tun
1: Visualisierung und Profilerstellung der normalen Verarbeitung 2: Einfaches Cython 3: Statische Typeinstellung durch cdef 4: Cythonisierung externer Module
Ausgangszustand
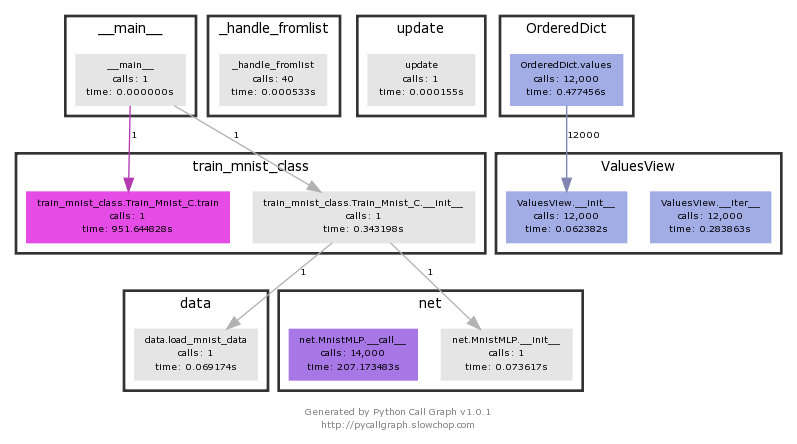
Die Visualisierung gibt Ihnen eine Vorstellung davon, was Zeit braucht. train_mnist.Train_Mnist.train Sie können sehen, dass es 951 Sekunden dauert.
Das Ergebnis eines normalen Profils ist unten.
ncalls: Anzahl der Anrufe tottime: Gesamtzeit, die diese Funktion verbringt percall: tottime geteilt durch ncalls cumtime: Gesamtzeit (von Anfang bis Ende) dieser Funktion, einschließlich untergeordneter Funktionen. Dieses Element wird auch in rekursiven Funktionen genau gemessen. percall: cumtime geteilt durch die Anzahl der primitiven Anrufe
Dieses Mal wurde der Code aus Gründen der Benutzerfreundlichkeit von Cython geändert, sodass sich die Verarbeitungszeit von der oben genannten unterscheidet. Ich wollte unbedingt Pycallgraph mit Cython verwenden, konnte es aber aufgrund meines Unwissens nicht verwenden. Wenn jemand weiß, wie man es benutzt, lass es mich wissen (die Verarbeitung des Cithon-Teils wird im normalen Gebrauch nicht aufgeführt).
Es endet in 755,154 Sekunden.
Ausführungsmethode
python -m cProfile
Profile.prof
37494628 function calls (35068627 primitive calls) in 755.154 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 448.089 0.006 448.651 0.006 adam.py:27(update_one_cpu)
114000 187.057 0.002 187.057 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 31.576 0.000 31.576 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 23.122 0.002 163.601 0.014 variable.py:216(backward)
Konzentrieren Sie sich basierend auf der 2: 8-Regel auf den Teil, dessen Verarbeitung am längsten dauert. Sie können sehen, dass adam.py fast den größten Teil der Verarbeitungszeit in Anspruch nimmt und die Matrixoperationen von numpy weiterhin die Verarbeitungszeit verwenden.
cython
Ich wollte es auch in Cython grafisch darstellen, aber ich hatte nicht genug Wissen über den Autor und konnte nicht nur den Verarbeitungsteil von Cython grafisch darstellen, also habe ich es profiliert.
Das Ergebnis ist langsamer als 800 Sekunden
Profile.prof
37466504 function calls (35040503 primitive calls) in 800.453 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 473.638 0.007 474.181 0.007 adam.py:27(update_one_cpu)
114000 199.589 0.002 199.589 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 33.706 0.000 33.706 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 24.754 0.002 173.816 0.014 variable.py:216(backward)
28000 9.944 0.000 10.392 0.000
Der Verarbeitungsteil von adam.py und variable.py ist langsamer als vor der Cythonisierung. Es besteht die Möglichkeit, dass es aufgrund der Zusammenarbeit zwischen der von Cython konvertierten c-Sprache und der externen Verarbeitung von Python langsam ist.
cdef
Ich habe cdef mit der Erwartung definiert, dass es schneller sein würde, einen statischen Typ im Voraus mit cdef zu definieren.
Vorbereitungen
Als ich es auf dem Mac verwendet habe, ist ein Fehler aufgetreten, und ich habe verschiedene Maßnahmen ergriffen.
Wenn ich versuche, cimport zu verwenden, wird der folgende Fehler angezeigt.
/Users/smap2/.pyxbld/temp.macosx-10.10-x86_64-3.4/pyrex/train_mnist_c2.c:242:10: fatal error: 'numpy/arrayobject.h' file not found
Im folgenden Verzeichnis
/usr/local/include/
Kopieren Sie das mit dem folgenden Befehl gefundene Header-Verzeichnis oder übergeben Sie es
find / -name arrayobject.h -print 2> /dev/null
Es waren 776 Sekunden.
Profile.prof
37466756 function calls (35040748 primitive calls) in 776.901 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 458.284 0.006 458.812 0.006 adam.py:27(update_one_cpu)
114000 194.834 0.002 194.834 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 33.120 0.000 33.120 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 24.025 0.002 168.772 0.014 variable.py:216(backward)
Es ist eine Verbesserung gegenüber der einfachen Cythonisierung, aber da sich in adam.py und variable.py nicht viel ändert, ist es aufgrund der zusätzlichen Verarbeitung der C-Sprache und der Python-Sprachkonvertierung langsamer als die Python-Verarbeitung.
adam.py cython
Ich habe versucht, den Teil zu beschleunigen, der die längste Verarbeitung benötigt, indem ich adam.py in cython konvertiert habe.
Infolgedessen zeigte es den Effekt, etwa 30 Sekunden schneller zu sein.
Profile.prof
37250749 function calls (34824741 primitive calls) in 727.414 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 430.495 0.006 430.537 0.006 optimizer.py:388(update_one)
114000 180.775 0.002 180.775 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 30.647 0.000 30.647 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 21.766 0.002 157.230 0.013 variable.py:216(backward)
Die Verarbeitungszeit von optimizer.py, einschließlich adam.py, ist etwa 20 Sekunden schneller als die von Python. Dies funktionierte und wurde schneller.
Zusammenfassung
Anstatt einfach zu versuchen, mit Cython zu beschleunigen, habe ich festgestellt, dass es eine Möglichkeit gibt, dass es effektiv ist, wenn ich es und Cython nur dort profiliere, wo es wirklich funktioniert. Es war ein Adventskalender, in dem ich erfahren konnte, dass der Zyklus von Hypothese, Visualisierung und Verifizierung wichtig ist, anstatt zu versuchen, ihn loszuwerden.
Der Code, den ich ausführen wollte, ist unten aufgeführt. https://github.com/SnowMasaya/chainer_cython_study
Referenz
Chainer: a neural network framework https://github.com/pfnet/chainer