[PYTHON] Nettoyage des données Comment gérer les valeurs manquantes et aberrantes
J'écrirai sur la gestion des valeurs aberrantes et des valeurs manquantes dans le nettoyage des données. Pensez-y comme fonctionnant sur Jupyter.
Préparation des données
Tout d'abord la préparation des données Vous pouvez facilement créer des données en utilisant la fonction make_classification de scikit-learn, alors préparez-les.
Référence: http://overlap.hatenablog.jp/entry/2015/10/08/022246
Lisons les données.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
data = pd.read_csv('2d_data.csv', header = None)
Les données préparées cette fois ressemblent à ceci
data = data.as_matrix()
data
#production
array([[ 1.00000000e+00, -7.42847611e+00, 1.50990301e+00],
[ 0.00000000e+00, 2.98069292e+00, 1.96082119e+00],
[ 0.00000000e+00, 3.98825476e+00, 4.63638899e+00],
[ 1.00000000e+00, -5.78943741e+00, -4.62161424e+00],
[ 1.00000000e+00, -4.89444674e+02, -3.26972997e+02],
[ 1.00000000e+00, -1.93394930e+00, -4.72763616e-02],
[ 0.00000000e+00, -1.61177146e+00, 5.93220121e+00],
[ 1.00000000e+00, -6.67015188e+00, nan],
[ 1.00000000e+00, -2.93141529e+00, -1.04474622e-01],
[ 0.00000000e+00, -7.47618437e-02, 1.07000182e+00],
[ 1.00000000e+00, -2.69179269e+00, 4.16877367e+00],
[ 0.00000000e+00, nan, 3.45343849e+00],
[ 0.00000000e+00, -1.35413500e+00, 3.75165665e+00],
[ 1.00000000e+00, -6.22947550e+00, -1.20943430e+00],
[ 0.00000000e+00, 2.77859414e+00, 7.58210258e+00],
[ 1.00000000e+00, -5.71957792e+00, -2.43509341e-01],
[ 0.00000000e+00, 9.28321714e-01, 3.20852039e+02],
[ 0.00000000e+00, 8.50475089e+01, 2.90895510e+00],
[ 1.00000000e+00, -6.02948927e+00, -1.83119942e+00],
[ 0.00000000e+00, 1.11602534e+00, 3.35360162e+00]])
Vous pouvez vérifier les valeurs aberrantes et manquantes (Nan). Divisons-le en données x et y.
X = data[:,1:3]
y = data[:,0].astype(int)
X.shape, y.shape
#production
((20, 2), (20,))
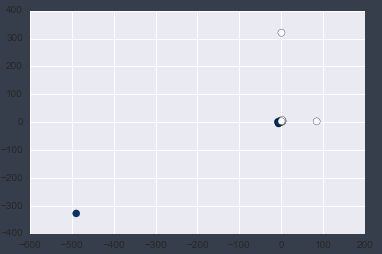
Maintenant, traçons les première et deuxième colonnes des données X.
plt.scatter(X[:, 0], X[:, 1], c=y, s=50, cmap='Blues');
Vous pouvez clairement voir les valeurs aberrantes en traçant.
Comment remplir les valeurs manquantes
Vous pouvez vérifier Nan en utilisant np.isnan (). S'il manque une valeur, elle renverra True.
np.isnan(X[:, 0]),np.isnan(X[:, 1])
#production
(array([False, False, False, False, False, False, False, False, False,
False, False, True, False, False, False, False, False, False,
False, False], dtype=bool),
array([False, False, False, False, False, False, False, True, False,
False, False, False, False, False, False, False, False, False,
False, False], dtype=bool))
Maintenant, créons "X1" et "y1" en excluant les valeurs manquantes.
X1 = X[~np.isnan(X[:, 1]) & ~np.isnan(X[:, 0])]
y1 = y[~np.isnan(X[:, 1]) & ~np.isnan(X[:, 0])]
X1, y1
#production
Out[139]:
(array([[ -7.42847611e+00, 1.50990301e+00],
[ 2.98069292e+00, 1.96082119e+00],
[ 3.98825476e+00, 4.63638899e+00],
[ -5.78943741e+00, -4.62161424e+00],
[ -4.89444674e+02, -3.26972997e+02],
[ -1.93394930e+00, -4.72763616e-02],
[ -1.61177146e+00, 5.93220121e+00],
[ -2.93141529e+00, -1.04474622e-01],
[ -7.47618437e-02, 1.07000182e+00],
[ -2.69179269e+00, 4.16877367e+00],
[ -1.35413500e+00, 3.75165665e+00],
[ -6.22947550e+00, -1.20943430e+00],
[ 2.77859414e+00, 7.58210258e+00],
[ -5.71957792e+00, -2.43509341e-01],
[ 9.28321714e-01, 3.20852039e+02],
[ 8.50475089e+01, 2.90895510e+00],
[ -6.02948927e+00, -1.83119942e+00],
[ 1.11602534e+00, 3.35360162e+00]]),
array([1, 0, 0, 1, 1, 1, 0, 1, 0, 1, 0, 1, 0, 1, 0, 0, 1, 0]))
Comment supprimer la valeur aberrante
X2 = X1[(abs(X1[:, 0] < 10)) & (abs(X1[:, 1]) < 10)]
y2 = y1[(abs(X1[:, 0] < 10)) & (abs(X1[:, 1]) < 10)]
Si vous l'écrivez comme ceci, il renverra un nombre de 10 ou plus comme suit.
(array([ True, True, True, True, True, True, True, True, True,
True, True, True, True, True, True, False, True, True], dtype=bool),
array([ True, True, True, True, False, True, True, True, True,
True, True, True, True, True, False, True, True, True], dtype=bool))
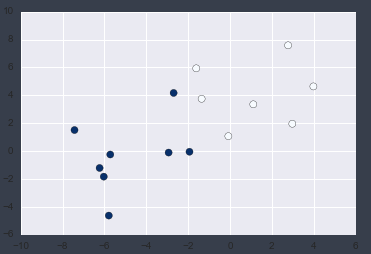
Supprimons les valeurs aberrantes.
X2 = X1[(abs(X1[:, 0] < 10)) & (abs(X1[:, 1]) < 10)]
y2 = y1[(abs(X1[:, 0] < 10)) & (abs(X1[:, 1]) < 10)]
Tracons-le.
plt.scatter(X2[:, 0], X2[:, 1],c = y2, s=50, cmap='Blues');
J'ai pu confirmer qu'il n'y avait pas de valeurs aberrantes!
Recommended Posts