[PYTHON] DBSCAN (Clustering) mit Scikit-Learn
Informationen zum Entwurf von DBSCAN, einem der Clustering-Algorithmen und zur einfachen Parameteroptimierung Ich schien keinen vollständigen japanischen Artikel zu haben, also schrieb ich ihn auf. Bitte beachten Sie, dass der Umriss von DBSCAN eine (grobe) japanische Übersetzung von [wikipedia] ist (https://en.wikipedia.org/wiki/DBSCAN).
Was ist DBSCAN?
- Abkürzung für "Dichtebasierte räumliche Clusterbildung von Anwendungen mit Rauschen"
- Einer der Clustering-Algorithmen
Überblick über den Algorithmus
-
- Klassifizieren Sie Punkte in 3
- Kernpunkt: Ein Punkt mit mindestens minPts benachbarter Punkte innerhalb eines Radius von ε
- Erreichbare Punkte (Randpunkte): Punkte, die nicht so viele benachbarte Punkte wie minPts innerhalb des Radius ε haben, aber Kernpunkte innerhalb des Radius ε
- Ausreißer: Punkte ohne benachbarte Punkte innerhalb des Radius ε
-
- Erstellen Sie einen Cluster aus einer Sammlung von Kernpunkten und weisen Sie jedem Cluster erreichbare Punkte zu.
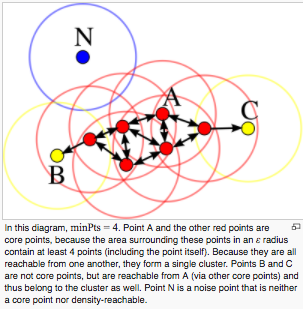
- Erstellen Sie einen Cluster aus einer Sammlung von Kernpunkten und weisen Sie jedem Cluster erreichbare Punkte zu.
[Aus Abbildung Wikipedia]
Vorteile
- Im Gegensatz zu k-means müssen Sie nicht zuerst die Anzahl der Cluster festlegen
- Kann auch in scharfen Clustern klassifiziert werden. Nimmt nicht an, dass der Cluster sphärisch ist
- Robust für Ausreißer.
- Es können zwei Parameter vorhanden sein, ε und minPts. Es ist auch einfach, den Parameterbereich zu bestimmen.
Nachteile
- Das Konzept der Grenzpunkte ist subtil und kann je nach Daten ändern, zu welchem Cluster es gehört.
- Die Genauigkeit variiert je nach Entfernungsberechnungsmethode.
- Es ist schwierig, ε und minPts richtig zu bestimmen, wenn die Daten dicht sind. In einigen Fällen werden die meisten Punkte in einem Cluster klassifiziert.
- Es ist schwierig, ε zu bestimmen, ohne die Daten zu kennen.
- (Nicht auf DBSCAN beschränkt) Wenn die Dimension wächst, wird sie vom Fluch der Dimension beeinflusst.
Unterschied zu anderen Algorithmen
Es ist eine Vergleichszahl jeder Methode in scikit-learn-Demoseite, aber die zweite von rechts ist DBSCAN. Sie können intuitiv erkennen, dass sie basierend auf der Dichte gruppiert sind.
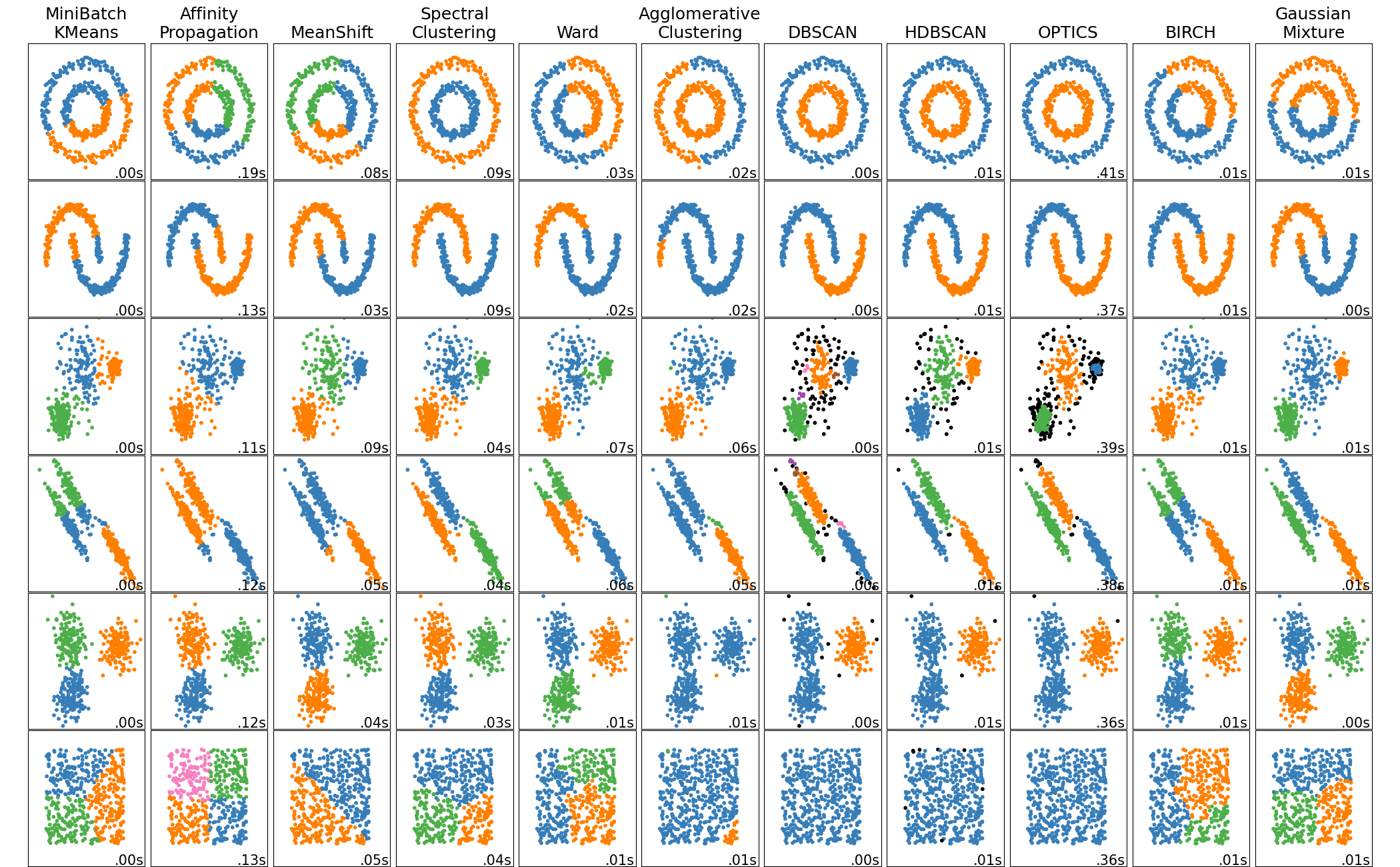
Abstimmung von ε und minPts
Wenn es zweidimensional ist, kann es visualisiert werden, um festzustellen, ob es gut klassifiziert ist. Wenn es jedoch dreidimensional oder mehr ist, ist es schwierig zu visualisieren und zu beurteilen. Der Ausreißer und die Anzahl der Cluster wurden wie folgt debuggt und angepasst. (Mit scikit-learn)
from sklearn.cluster import DBSCAN
for eps in range(0.1,3,0.1):
for minPts in range(1,20):
dbscan = DBSCAN(eps=eps,min_samples=minPts).fit(X)
y_dbscan = dbscan.labels_
print("eps:",eps,",minPts:", minPts)
#Anzahl der Ausreißer
print(len(np.where(y_dbscan ==-1)[0]))
#Anzahl der Cluster
print(np.max(y_dbscan)))
#Anzahl der Punkte in Cluster 1
print(len(np.where(y_dbscan ==0)[0]))
#Anzahl der Punkte in Cluster 2
print(len(np.where(y_dbscan ==1)[0]))
DBSCAN-bezogene Links
- wikipedia
- https://en.wikipedia.org/wiki/DBSCAN
- Offizielle Dokumentation
- http://scikit-learn.org/stable/modules/generated/sklearn.cluster.DBSCAN.html
- Offizielle Demoseite
- http://scikit-learn.org/stable/auto_examples/cluster/plot_dbscan.html
- Qiita
- http://qiita.com/ynakayama/items/46d5f2e49f57c7de98a4
- http://qiita.com/yamaguchiyuto/items/82c57c5d44833f5a33c7
Nachtrag
Japanische Version von Wikipedia wurde mit zusätzlichen Beschreibungen aktualisiert. Es ist leicht zu verstehen.
Recommended Posts