Visualisieren Sie Netzwerkdaten aus IPython Notebook mit Cytoscape Part 1
Einführung
Der neulich veröffentlichte Artikel wurde mit dem Schwerpunkt geschrieben, wie Workshops mit Datenvisualisierungssoftware effizient durchgeführt werden können. Ich möchte den Inhalt des eigentlichen Workshops, den ich ein wenig vorgestellt habe, dh den Teil des Verfahrens zur interaktiven Visualisierung des Netzwerks, auch auf Japanisch vorstellen.
Hintergrund des Problems
Unabhängig von der Netzwerkanalyse nimmt die Anzahl der Tools zu, die für die Datenanalyse und -visualisierung verwendet werden können, aber die Menge an Informationen, die durch deren Kombination tatsächlich verwendet werden können, ist immer noch gering. In letzter Zeit sind Python und R zum Standard in den Bereichen Datenanalyse und -visualisierung geworden. Hier einige Beispiele für die Verwendung von Python. Verwenden Sie die von uns entwickelte Netzwerkvisualisierungssoftware Cytoscape, NetworkX usw. Es ist ein Versuch, die zahlreichen Analysefunktionen von Cytoscape und die flexible Visualisierungsfunktion von Cytoscape zu "nutzen". Der Artikel auf Englisch ist hier, aber ich hoffe, dass Japaner populärere Kombinationen dieser Tools finden. Ich denke, es ist einfach, aber ich werde es vorstellen.
Umgebungseinstellung
Es ist am einfachsten, mit Docker fortzufahren. Die meisten Leser dieses Artikels sind Programmierer, daher werde ich die Details weglassen, aber das folgende Verfahren wird die gleiche Umgebung, die ich verwendet habe, vollständig reproduzieren. Natürlich können Sie Ihre eigene Umgebung lokal erstellen, aber ich denke, es wird zusätzliche Anstrengungen erfordern.
Erforderliche Software
- Git
- Docker (getestet mit 1.5)
Dies ist in Ordnung, wenn Sie nur die Grundlagen der Notiz ausprobieren möchten, das Netzwerk jedoch mit Cytoscape visualisieren möchten, müssen Sie es ebenfalls installieren. Die notwendige Software in diesem Fall ist
- Java 8 neueste Version
- Cytoscape 3.2.1
- cyREST App - Ein Add-On (App genannt), das Cytoscape eine REST-API hinzufügt. Sie können direkt über die Schaltfläche Installieren installieren, indem Sie diese Seite (http://apps.cytoscape.org/apps/cyrest) öffnen, während Cytoscape ausgeführt wird. Die neueste Version (0.9.13 oder höher) ist erforderlich.
Erstellen einer Notebook-Umgebung
Grundsätzlich wird alles automatisch mit folgendem Befehl erledigt:
git clone https://github.com/idekerlab/vizbi-2015
cd vizbi-2015
docker run -d -p 80:8888 -v $PWD:/notebooks -e "PASSWORD=(PW für den Zugriff auf Notebooks)" -e "USE_HTTP=1" idekerlab/vizbi-2015
Wenn es ausgeführt wird, müssen Sie nur über Ihren Browser darauf zugreifen. Wenn Sie boot2docker verwenden, sollte die Adresse folgendermaßen aussehen:
http://192.168.59.103/

Jetzt können Sie sofort auf die Notizbücher in tutorials zugreifen

Wenn Sie das IPython-Notizbuch überhaupt nicht berührt haben, ist es hilfreich, einige grundlegende Notizoperationen und die Verwendung eines speziellen Befehls namens magic in Lektion 0 zu lernen.
Leider sind alle diese Notizbücher auf Englisch, daher werde ich einige Kommentare auf Japanisch hinzufügen.
Was ist cyREST?

Cytoscape ist eine Open-Source-Software zur Netzwerkanalyse und -visualisierung, die in der Biologie weit verbreitet ist. Da es sich um eine GUI-basierte Operation handelt, ist die Installation relativ einfach, aber für diejenigen, die sie schwer verwenden, gibt es einige Leute, die die Verwendung schwierig finden, da es keine Mittel zur Automatisierung gibt. Um dieses Problem zu lösen, wurde eine weit verbreitete RESTful-API mit der ressourcenorientierten Entwurfsmethode, einer neueren Standardmethode, und App erstellt. Es ist implementiert als.
Beim Zugriff aus dem Docker-Container auf Boot2docker
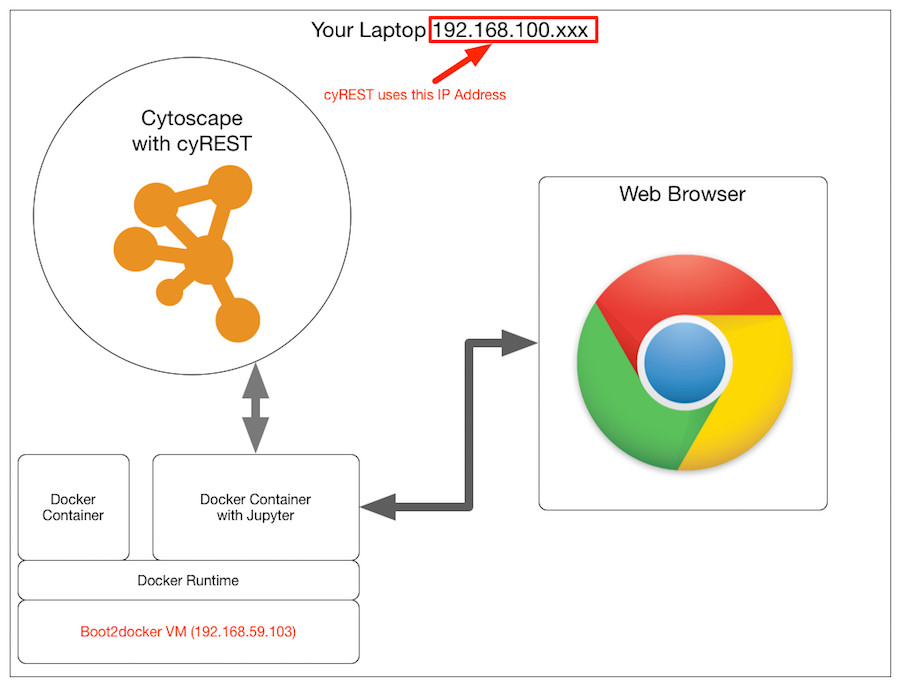
Es ist in Ordnung, wenn Sie alles direkt auf Ihrem lokalen Computer installieren, aber nicht über den Container auf boot2docker mit localhost darauf zugreifen können. Daher müssen Sie die IP-Adresse des Computers angeben, auf dem Cytoscape ausgeführt wird, und am Anfang des Notebooks Sie benötigen folgenden Code:
#IP-Adresse des cyREST-Servers (die Adresse des Computers, auf dem Cytoscape ausgeführt wird). Muss eingestellt werden!
IP = '10.0.1.6'
#cyREST-Serverinformationen. Der Standardport ist 1234
PORT_NUMBER = 1234
#Speichern Sie die Basis-URL als Konstante
BASE = 'http://' + IP + ':' + str(PORT_NUMBER) + '/v1/'
Tatsächlicher Fluss
Dieses Mal werde ich ein Beispiel hochladen, das sehr grundlegende Funktionen als Notizbuch verwendet.
Netzwerkgenerierung und POST
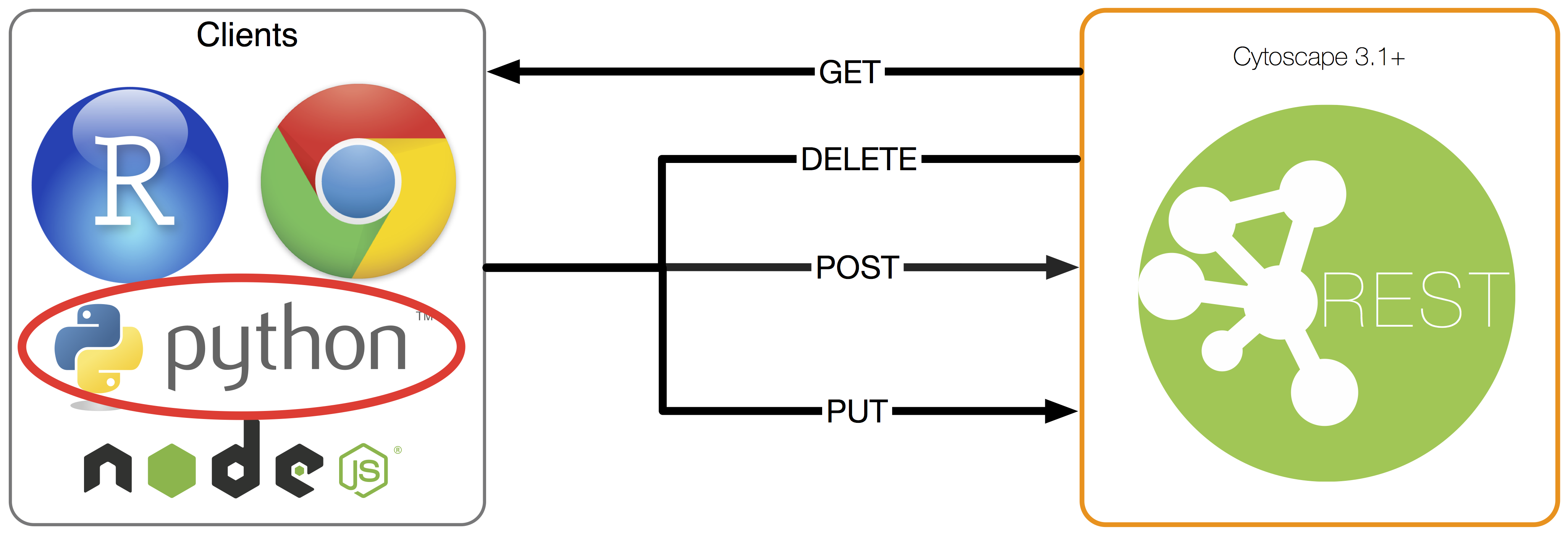
Die Funktion von cyREST ist so einfach wie das Aktivieren von [CRUD] -Operationen (http://ja.wikipedia.org/wiki/CRUD) für die in Cytoscape verwendeten grundlegenden Datenstrukturen. ist. Ich werde hier nicht auf Details eingehen, aber es unterstützt grundlegende CRUD für die folgenden Daten:
- CyNetwork
- Group
- CyTable
- Visual Style
Weitere Informationen finden Sie in der offiziellen Cytoscape-Dokumentation. Wir fügen derzeit Funktionen hinzu und beheben Fehler für die Version 1.0.0, sodass einige Funktionen zur endgültigen Version hinzugefügt werden.
Ich denke, es ist schneller, den Code tatsächlich zu betrachten, um die Grundfunktionalität zu nutzen. Hier ist ein Beispiel. Der folgende Code transformiert die von NetworkX generierten Daten in JSON in einem von Cytoscape akzeptierten Format und POSTs.
#Anzahl der Knoten im generierten Netzwerk
NUM_NODES = 100
#Erstellen Sie ein kleines Netzwerk ohne Skalierung
scale_free_graph = nx.scale_free_graph(NUM_NODES)
#Nennen Sie es als Netzwerkattribut
scale_free_graph.graph['name'] = 'Skalierungsfreies Netzwerk(Anzahl der Knoten: ' + str(NUM_NODES) + ')'
#Cytoscape es mit einem Dienstprogramm.Holen Sie sich ein Wörterbuchobjekt, das in das JSON-Format JSON konvertiert werden kann
cy_netwiork1 = cy.from_networkx(scale_free_graph)
#POST-Netzwerk zu Cytoscape(Create)
res = requests.post(BASE + 'networks', data=json.dumps(cy_netwiork1), headers=HEADERS)
#SUID des generierten Netzwerks(Session-Unique ID)Erhalten
suid = res.json()['networkSUID']
#Verwenden Sie diese SUID, um einen automatischen Layoutalgorithmus auszuführen (hier ein Federmodell).
requests.get(BASE + 'apply/layouts/force-directed/' + str(suid))
#Voreingestellter visueller Stil(Siehe unten)Anwenden
requests.get(BASE + 'apply/styles/Directed/' + str(suid))
#Die eingebettete Visualisierung führt zu diesem Hinweis mithilfe der Bild-API
Image(url=BASE+'networks/' + str(suid) + '/views/first.png', embed=True)
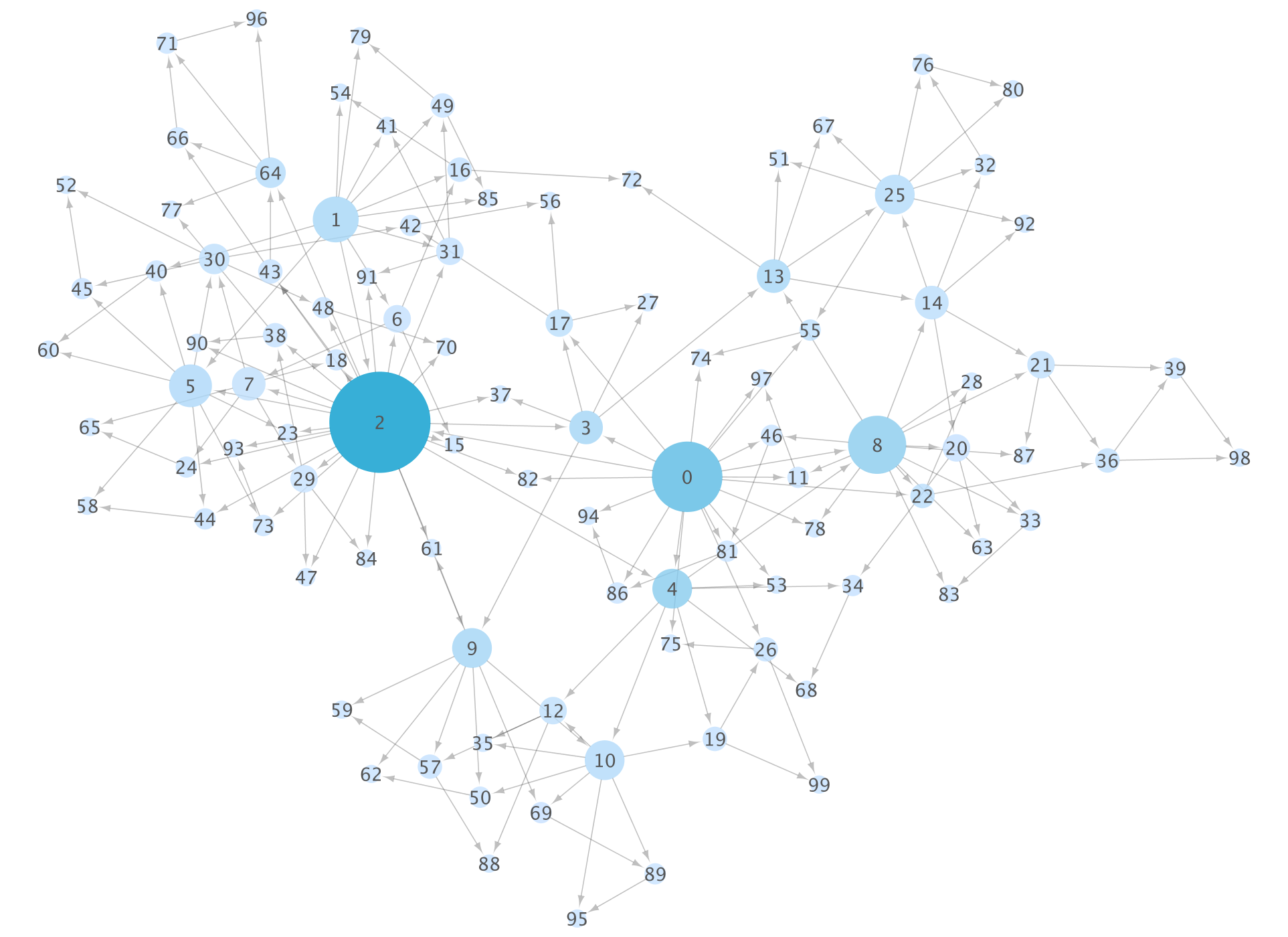
Wenn Sie dies tun, hat die Notizenseite ein Bild wie dieses (das ein zufälliges Netzwerk erzeugt, sodass sich die Topologie bei jedem Lauf ändert):

Und das Gleiche wird auf der Cytoscape-Seite angezeigt.

Diese Kombination ist praktisch, da Sie mithilfe der __ (Netzwerk-) Datenanalyse und -visualisierung selektiv auswählen können, wie viel automatisiert und was interaktiv untersucht werden soll. Mit anderen Worten, Daten können einfach und automatisch durch den in das Notizbuch geschriebenen Python-Code analysiert und bei Bedarf über die interaktive Cytoscape-Benutzeroberfläche bedient werden.
Wenn Sie dieselben Daten visualisieren, die die Grundfunktionen von NetworkX verwenden, sieht dies übrigens wie folgt aus:

Wenn Sie dies nur mit NetworkX tun, schreiben Sie von hier aus verschiedene Parameter als Code auf, aber es ist oft einfacher, einige Arbeiten (Korrektur des feinen Layouts usw.) von Hand auszuführen.
Es wird empfohlen, in einer Umgebung mit zwei Monitoren zu arbeiten, da Sie bei dieser Technik häufig zwischen den beiden Fenstern wechseln.
Verwenden Sie die grundlegende Visualisierungsfunktion (Visual Style).
Visualisierung ist eine Kernfunktion von Cytoscape, aber sehr einfach. das ist,
__ Geben Sie eine Funktion an, die Daten visuellen Variablen zuordnet und zur Visualisierung verwendet. Wenn die Funktion nicht vorhanden ist, verwenden Sie den im Voraus angegebenen Standardwert __
Es wird genannt. Ersteres heißt Visual Mapping Functions, letzteres heißt defaults und ihre Menge heißt Visual Style. Dieses Mal werde ich nur einen kleinen Teil vorstellen, aber ich werde verschiedene Werte anpassen und den visuellen Stil auf der __Cytoscape-Seite manuell als JSON mit der GET-Methode bearbeiten lassen. Auf dieser Grundlage werde ich den neuen Stil als Code verwenden Ich denke, die Abkürzung zum Erlernen des Grundkonzepts besteht darin, __ zu erstellen.
PUT (Update) den Wert der Standardeinstellungen
Dieser Teil ist sehr einfach. Sie müssen lediglich eine Liste der Farben und Formen erstellen, die verwendet werden, wenn die Zuordnung nicht vorhanden ist, und anschließend PUT.
defaults = [
{
'visualProperty':'NODE_FILL_COLOR',
'value': '#FFFFFF'
},
{
'visualProperty':'NODE_BORDER_WIDTH',
'value': 0
},
{
'visualProperty':'NODE_LABEL_FONT_SIZE',
'value': 18
},
{
'visualProperty':'NODE_LABEL_COLOR',
'value': '#555555'
},
{
'visualProperty':'NODE_SIZE',
'value': 30
},
{
'visualProperty': 'EDGE_WIDTH',
'value': 1
},
{
'visualProperty': 'NODE_TRANSPARENCY',
'value': 200
},
{
'visualProperty': 'EDGE_TRANSPARENCY',
'value': 80
}
]
res = requests.put(BASE + 'styles/Directed/defaults', data=json.dumps(defaults), headers=HEADERS)
Mapping generieren
NetworkX verfügt über viele Algorithmen zur Diagrammanalyse, mit denen verschiedene Indikatoren berechnet werden können. Sie können die flexible visuelle Zuordnungsfunktion von Cytoscape (http://wiki.cytoscape.org/Cytoscape_3/UserManual/Styles) verwenden, um solche Zahlen visuellen Eigenschaften zuzuordnen.
#Ordnen Sie die Knotenreihenfolge der Größe zu
node_size_mapping = {
'mappingType': 'continuous',
'mappingColumn': 'degree',
'mappingColumnType': 'Double',
'visualProperty': 'NODE_SIZE',
'points':[
{
'value': 1,
'lesser': '15',
'equal': '15',
'greater': '15'
},
{
'value': max(degree.values()),
'lesser': '100',
'equal': '100',
'greater': '100'
}
]
}
#Zuordnung zwischen Knotenfarbe
node_color_mapping = {
'mappingType': 'continuous',
'mappingColumn': 'betweenness',
'mappingColumnType': 'Double',
'visualProperty': 'NODE_FILL_COLOR',
'points':[
{
'value': 0,
'lesser': '#C6E2FF',
'equal': '#C6E2FF',
'greater': '#C6E2FF'
},
{
'value': max(bc.values()),
'lesser': '#009ACD',
'equal': '#009ACD',
'greater': '#009ACD'
}
]
}
res = requests.post(BASE + 'styles/Directed/mappings', data=json.dumps([node_size_mapping, node_color_mapping]), headers=HEADERS)
In diesem Notebook-Beispiel wird möglicherweise ein Desktop wie dieser konfiguriert

Von hier aus können Sie die Visualisierung manuell weiter optimieren oder den Code auf der Notebook-Seite ändern, um denselben Workflow auf andere Daten anzuwenden.
abschließend
Dieser Artikel sieht aus wie eine Digest-Version des Workshops, der neulich abgehalten wurde. Wenn Sie also an den Details interessiert sind, ist er auf Englisch [hier](https://github.com/idekerlab/cyREST] verfügbar. Bitte folgen Sie aus / wiki / VIZBI-2015-Tutorial).
Ich möchte zu einem späteren Zeitpunkt kompliziertere Beispiele veröffentlichen.
Recommended Posts
