Visualisez les données réseau à partir d'IPython Notebook à l'aide de Cytoscape Partie 1
introduction
Article posté l'autre jour a été rédigé en mettant l'accent sur la manière de procéder efficacement aux ateliers en utilisant un logiciel de visualisation de données. , Je voudrais présenter le contenu de l'atelier proprement dit que j'ai un peu introduit en ce sens, c'est-à-dire la partie de la procédure de visualisation interactive du réseau, en japonais également.
Contexte du problème
Indépendamment de l'analyse de réseau, le nombre d'outils pouvant être utilisés pour l'analyse et la visualisation des données augmente, mais la quantité d'informations qui peuvent être réellement utilisées en les combinant est encore faible. Récemment, Python et R sont devenus la norme dans les domaines de l'analyse et de la visualisation de données, mais voici quelques exemples utilisant Python. En utilisant le logiciel de visualisation réseau appelé Cytoscape que nous développons, NetworkX etc. Il s'agit d'une tentative de «profiter» des nombreuses fonctions d'analyse de Cytoscape et de la fonction de visualisation flexible de Cytoscape. L'article en anglais est ici, mais j'espère que les Japonais trouveront des combinaisons plus populaires de ces outils. Je pense que c'est facile, mais je vais vous le présenter.
Cadre environnemental
Il est plus simple de procéder avec Docker. La plupart des lecteurs de cet article sont des programmeurs, je vais donc omettre les détails, mais la procédure suivante reproduira complètement le même environnement que j'ai utilisé. Bien sûr, vous pouvez créer votre propre environnement localement, mais je pense que cela demandera des efforts supplémentaires.
Logiciel requis
- Git
- Docker (testé avec 1.5)
Cela convient si vous souhaitez simplement essayer les parties de base de la note, mais si vous souhaitez visualiser le réseau à l'aide de Cytoscape, vous devez également l'installer. Le logiciel nécessaire dans ce cas est
- Dernière version de Java 8
- Cytoscape 3.2.1
- cyREST App - Un module complémentaire (appelé App) qui ajoute une API REST à Cytoscape. Vous pouvez installer directement à partir du bouton Installer en ouvrant cette page (http://apps.cytoscape.org/apps/cyrest) pendant que Cytoscape est en cours d'exécution. La dernière version (0.9.13 ou ultérieure) est requise.
Créer un environnement de notebook
En gros, tout se fait automatiquement avec la commande suivante:
git clone https://github.com/idekerlab/vizbi-2015
cd vizbi-2015
docker run -d -p 80:8888 -v $PWD:/notebooks -e "PASSWORD=(PW pour accéder aux notebooks)" -e "USE_HTTP=1" idekerlab/vizbi-2015
S'il est en cours d'exécution, il vous suffit d'y accéder depuis votre navigateur. Si vous utilisez boot2docker, l'adresse devrait ressembler à ceci:
http://192.168.59.103/

Vous pouvez désormais accéder immédiatement aux blocs-notes dans tutoriels

Si vous n'avez pas du tout touché au bloc-notes IPython, il sera utile d'apprendre certaines opérations de base sur les notes et comment utiliser une commande spéciale appelée magic dans la leçon 0.
Malheureusement, tous ces cahiers sont en anglais, je vais donc ajouter quelques commentaires en japonais.
Qu'est-ce que cyREST?

Cytoscape est un logiciel open source d'analyse et de visualisation de réseau largement utilisé dans la communauté de la biologie. Comme il s'agit d'une opération basée sur l'interface graphique, elle est relativement facile à installer, mais pour ceux qui l'utilisent dur, certaines personnes trouvent cela difficile à utiliser car il n'y a aucun moyen d'automatisation. Pour résoudre ce problème, une API RESTful largement utilisée a été créée en utilisant la méthode de conception orientée ressources, qui est une méthode standard récente, et App. Il est implémenté comme.
Lors de l'accès depuis le conteneur Docker sur Boot2docker
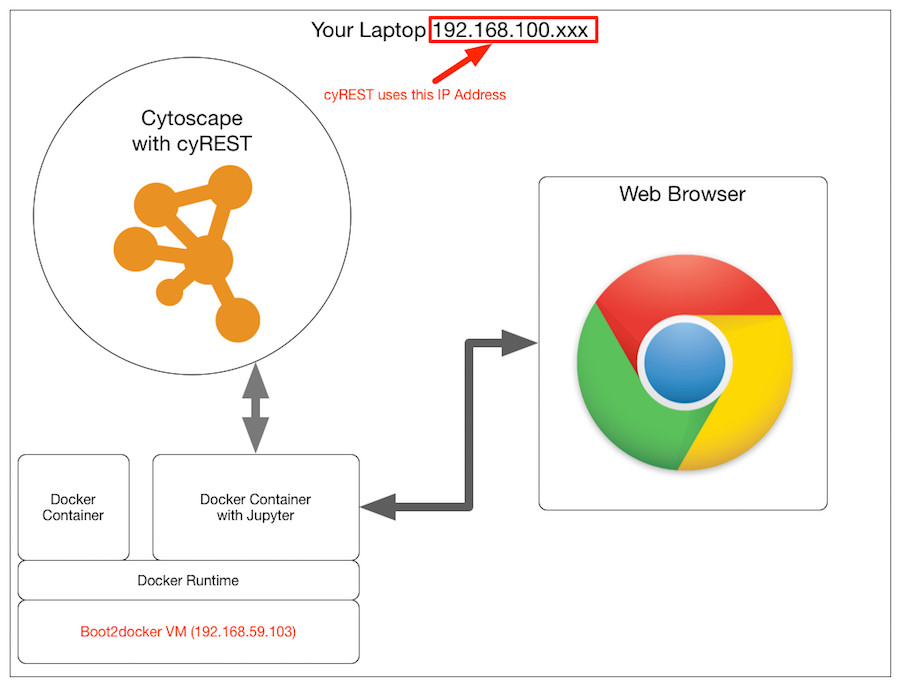
Ce n'est pas grave si vous installez tout directement sur votre machine locale, mais vous ne pouvez pas y accéder à partir du conteneur sur boot2docker avec localhost, vous devez donc spécifier l'adresse IP de la machine sur laquelle Cytoscape est en cours d'exécution, et au début du notebook Vous aurez besoin d'un code comme celui-ci:
#Adresse IP du serveur cyREST (l'adresse de la machine exécutant Cytoscape). Doit être réglé!
IP = '10.0.1.6'
#Informations sur le serveur cyREST. Le port par défaut est 1234
PORT_NUMBER = 1234
#Enregistrez l'URL de base en tant que constante
BASE = 'http://' + IP + ':' + str(PORT_NUMBER) + '/v1/'
Débit réel
Cette fois, je vais télécharger un exemple qui utilise des fonctions très basiques comme cahier.
Génération de réseau et POST
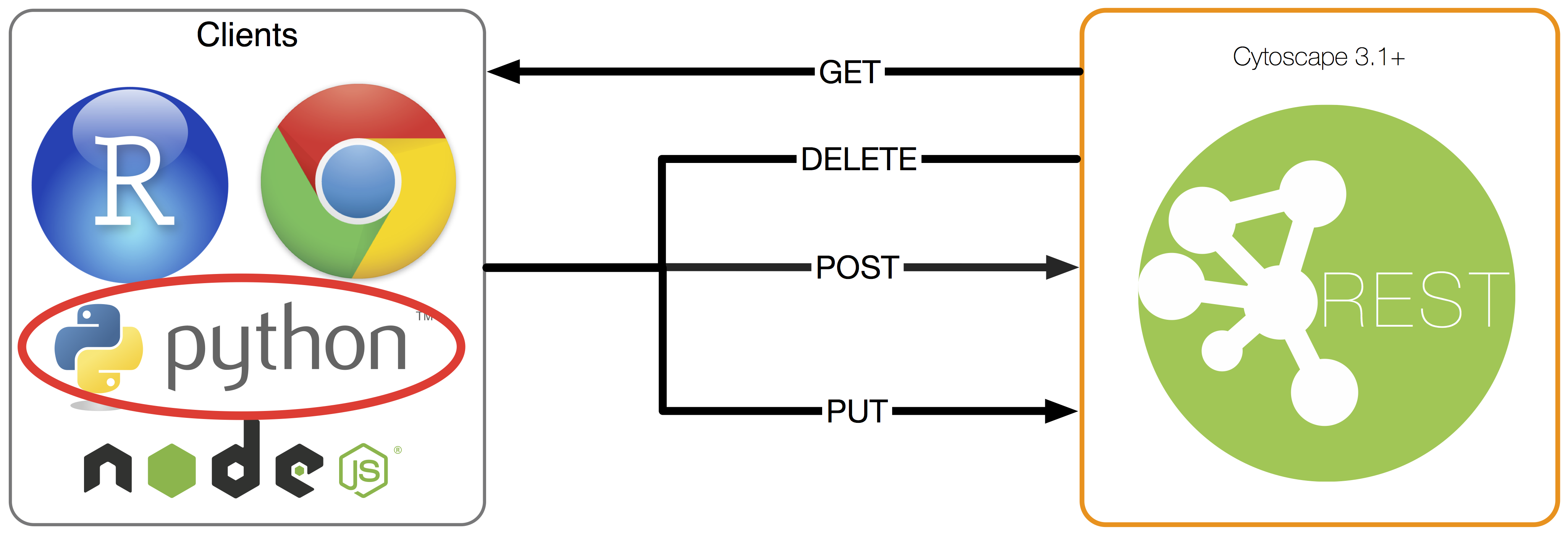
La fonction de cyREST est aussi simple que d'activer les opérations CRUD sur les structures de données de base utilisées dans Cytoscape. est. Je n'entrerai pas dans les détails ici, mais il prend en charge CRUD de base pour les données suivantes:
- CyNetwork
- Group
- CyTable
- Visual Style
Consultez la documentation officielle de Cytoscape pour plus de détails. Nous ajoutons actuellement des fonctionnalités et corrigeons des bugs pour la version v1.0.0, donc certaines fonctionnalités seront ajoutées à la version finale.
Je pense qu'il est plus rapide de regarder le code pour utiliser les fonctionnalités de base, alors voici un exemple. Le code ci-dessous transforme les données générées par NetworkX en JSON dans un format accepté par Cytoscape et le POST.
#Nombre de nœuds dans le réseau généré
NUM_NODES = 100
#Créez un réseau sans échelle à petite échelle
scale_free_graph = nx.scale_free_graph(NUM_NODES)
#Nommez-le comme un attribut de réseau
scale_free_graph.graph['name'] = 'Réseau sans échelle(Nombre de nœuds: ' + str(NUM_NODES) + ')'
#Cytoscape avec un utilitaire.Obtenez un objet dictionnaire qui peut être converti au format js JSON
cy_netwiork1 = cy.from_networkx(scale_free_graph)
#Réseau POST sur Cytoscape(Create)
res = requests.post(BASE + 'networks', data=json.dumps(cy_netwiork1), headers=HEADERS)
#SUID du réseau généré(Session-Unique ID)Avoir
suid = res.json()['networkSUID']
#Utilisez ce SUID pour exécuter un algorithme de mise en page automatique (ici, un modèle à ressort)
requests.get(BASE + 'apply/layouts/force-directed/' + str(suid))
#Style visuel prédéfini(Voir ci-dessous)Appliquer
requests.get(BASE + 'apply/styles/Directed/' + str(suid))
#Incorporer les résultats de visualisation dans cette note à l'aide de l'API d'image
Image(url=BASE+'networks/' + str(suid) + '/views/first.png', embed=True)
Lorsque vous faites cela, le côté note aura une image comme celle-ci (qui génère un réseau aléatoire, donc la topologie changera à chaque exécution):

Et la même chose est affichée du côté de Cytoscape.

Cette combinaison est pratique car vous pouvez utiliser de manière sélective le travail d'analyse et de visualisation des données __ (réseau) pour choisir combien d'automatisation et quoi faire de l'exploration interactive. En d'autres termes, les données peuvent être facilement et automatiquement analysées par le code Python écrit dans le cahier, et peuvent être exploitées à partir de l'interface graphique interactive Cytoscape selon les besoins.
À propos, si vous visualisez les mêmes données que celles utilisées avec les fonctions de base de NetworkX, ce sera comme suit:

Lorsque vous le faites uniquement avec NetworkX, divers paramètres sont écrits sous forme de code à partir d'ici, mais il est souvent plus facile de faire du travail (correction de la mise en page fine, etc.) à la main.
Il est recommandé de travailler dans un environnement à deux moniteurs car vous basculerez souvent entre les deux fenêtres lors de l'utilisation de cette technique.
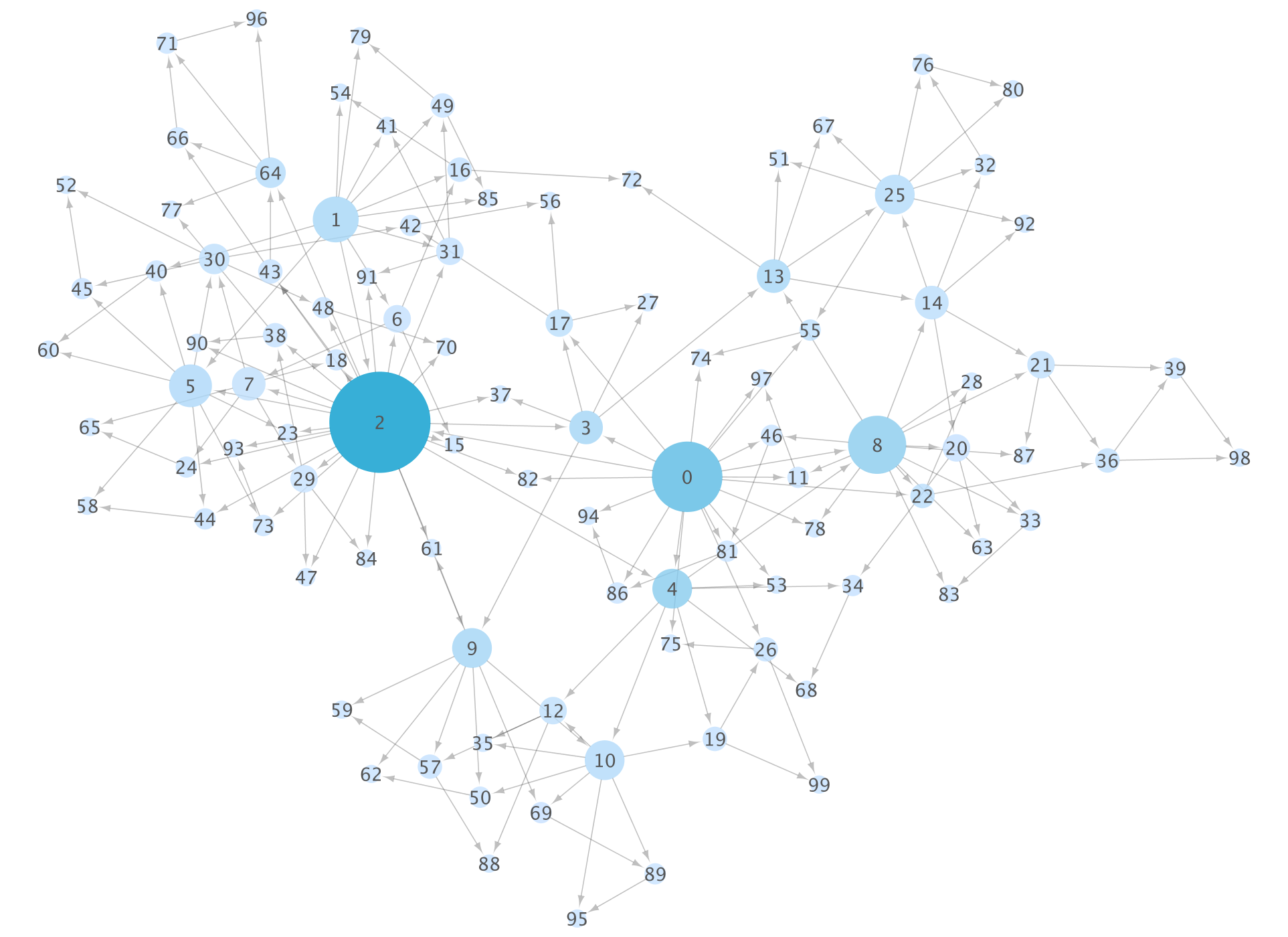
Utiliser la fonction de visualisation de base (style visuel)
La visualisation est une fonctionnalité essentielle de Cytoscape, mais elle est très simple à faire. C'est,
__ Spécifiez une fonction qui mappe les données sur des variables visuelles et utilisez-la pour la visualisation. Si la fonction n'existe pas, utilisez la valeur par défaut spécifiée à l'avance __
On l'appelle. La première s'appelle Visual Mapping Functions, la seconde s'appelle defaults et leur ensemble s'appelle Visual Style. Cette fois, je n'introduirai qu'une petite partie, mais j'ajusterai diverses valeurs et j'obtiendrai le style visuel manuellement édité du côté __Cytoscape en tant que JSON avec la méthode GET, et sur cette base, j'utiliserai le nouveau style comme code Je pense que le raccourci pour apprendre le concept de base est de faire le travail de création de __.
PUT (mettre à jour) la valeur des valeurs par défaut
Cette partie est très simple. Tout ce que vous avez à faire est de créer une liste des couleurs et des formes qui seront utilisées si le mappage n'existe pas, puis PUT.
defaults = [
{
'visualProperty':'NODE_FILL_COLOR',
'value': '#FFFFFF'
},
{
'visualProperty':'NODE_BORDER_WIDTH',
'value': 0
},
{
'visualProperty':'NODE_LABEL_FONT_SIZE',
'value': 18
},
{
'visualProperty':'NODE_LABEL_COLOR',
'value': '#555555'
},
{
'visualProperty':'NODE_SIZE',
'value': 30
},
{
'visualProperty': 'EDGE_WIDTH',
'value': 1
},
{
'visualProperty': 'NODE_TRANSPARENCY',
'value': 200
},
{
'visualProperty': 'EDGE_TRANSPARENCY',
'value': 80
}
]
res = requests.put(BASE + 'styles/Directed/defaults', data=json.dumps(defaults), headers=HEADERS)
Générer un mappage
NetworkX possède de nombreux algorithmes d'analyse graphique, qui peuvent être utilisés pour calculer divers indicateurs. Vous pouvez utiliser la fonction flexible de cartographie visuelle de Cytoscape (http://wiki.cytoscape.org/Cytoscape_3/UserManual/Styles) pour mapper ces nombres sur des propriétés visuelles.
#Mapper l'ordre des nœuds en magnitude
node_size_mapping = {
'mappingType': 'continuous',
'mappingColumn': 'degree',
'mappingColumnType': 'Double',
'visualProperty': 'NODE_SIZE',
'points':[
{
'value': 1,
'lesser': '15',
'equal': '15',
'greater': '15'
},
{
'value': max(degree.values()),
'lesser': '100',
'equal': '100',
'greater': '100'
}
]
}
#Mapper l'intervalle à la couleur du nœud
node_color_mapping = {
'mappingType': 'continuous',
'mappingColumn': 'betweenness',
'mappingColumnType': 'Double',
'visualProperty': 'NODE_FILL_COLOR',
'points':[
{
'value': 0,
'lesser': '#C6E2FF',
'equal': '#C6E2FF',
'greater': '#C6E2FF'
},
{
'value': max(bc.values()),
'lesser': '#009ACD',
'equal': '#009ACD',
'greater': '#009ACD'
}
]
}
res = requests.post(BASE + 'styles/Directed/mappings', data=json.dumps([node_size_mapping, node_color_mapping]), headers=HEADERS)
Dans cet exemple de notebook, un bureau comme celui-ci sera finalement configuré

À partir de là, vous pouvez affiner davantage la visualisation manuellement, ou vous pouvez modifier le code du côté du bloc-notes pour appliquer le même flux de travail à d'autres données.
en conclusion
Cet article ressemble à une version condensée de l'atelier qui a eu lieu l'autre jour, donc si vous êtes intéressé par les détails, la version anglaise est [ici](https://github.com/idekerlab/cyREST] Veuillez suivre de / wiki / VIZBI-2015-Tutorial).
J'aimerais publier des échantillons plus compliqués à une date ultérieure.
Recommended Posts
