[PYTHON] Solution de première place du concours national d'IA médicale 2020
0. Introduction
J'ai remporté le concours national d'IA médicale 2020, un concours de données de table médicale parrainé par la AI & Machine Learning Society (AIMS) de l'Université d'Osaka (1ère place, top 6%), je publierai donc la solution.
Comme vous le savez peut-être d'après les articles jusqu'à présent, je ne suis pas très bon à la compétition de données de table parce que je suis spécialisé dans les données d'image. Veuillez noter qu'il peut y avoir des erreurs et des parties irrationnelles.
En outre, cet article est essentiellement écrit pour ceux qui ont participé au concours. Il n'y a aucune explication sur les noms de colonnes et les quantités de caractéristiques spécifiques, vous pouvez donc ignorer ces parties. (La partie technique est expliquée en détail!)
De même, puisqu'il s'agissait d'un concours privé, vous ne pouvez accéder au site du concours que les participants.
1. À propos du concours
1-1. Aperçu du concours
La compétition est une compétition de données de table pour prédire la mortalité des patients COVID-19. Nous analyserons les données sur les facteurs de risque (localisation, présence ou absence de pneumonie, âge, etc.) pouvant conduire à un décès donné, et créerons et soumettons un modèle. La période de compétition est très courte, 2 jours (entre le 26/09, 14h00 et le 27/09, 12h00) et la clé est la rapidité avec laquelle l'inférence, la mise en œuvre et l'optimisation peuvent être effectuées. .. De plus, la participation au concours était limitée aux étudiants.
1-2. Index d'évaluation
L'indice d'évaluation est la zone sous la courbe ROC. Cet indice d'évaluation a déjà été expliqué dans l'article suivant, je vais donc l'omettre. [Melanoma competition-Area under ROC curve](https://qiita.com/tachyon777/items/05e7d35b7e0b53ef03dd#1-2%E8%A9%95%E4%BE%A1%E6%8C%87%E6%A8% 99)
1-3. Ensemble de données
L'ensemble de données est simple ・ Train.csv ・ Test.csv ・ Sample_submission.csv Il se compose de trois parties. Apprenez avec former, prédire le test et soumettre sous la forme de sample_submission.
Concernant le contenu des données, il y avait les éléments suivants. ・ Adresse du patient (détails) ·âge ・ Emplacement de l'hôpital ・ Lieu de naissance du patient ・ Installations où les patients ont été traités ・ Avez-vous des antécédents de contact avec d'autres patients COVID? ・ Résultats des tests PCR ·pneumonie ・ Date d'apparition ・ Date d'hospitalisation (date de consultation) ・ Intubation ・ Hospitalisation ou ambulatoire ・ Présence ou absence d'insuffisance rénale chronique, de diabète, d'hypertension artérielle, de maladies cardiovasculaires, d'asthme et d'autres maladies sous-jacentes
Étant donné que le résultat de la prédiction de test.csv est en fait reflété dans LeaderBoard (et non dans Code Competition), la quantité donnée en tant que données de test est importante et on pensait que des méthodes telles que le pseudo-étiquetage seraient efficaces.
2.EDA Je n'avais rien à faire. C'est parce qu'Akiyama, qui dirige le concours, a sorti un cahier combinant EDA et base au début de la compétition, et presque toutes les informations nécessaires étaient disponibles. En particulier, le score de feature_importance de LightGBM a été répertorié, et le reste était juste pour implémenter le modèle. Grâce à une analyse de données très simple à comprendre, j'ai pu commencer à mettre en œuvre le modèle immédiatement.
3. Théorie
En théorie, j'expliquerai cette fois la méthode clé de ma solution.
3-1. Target Encoding Le codage cible est une méthode d'utilisation de la «valeur moyenne de l'étiquette correcte de la catégorie» elle-même comme quantité de caractéristiques dans la variable de catégorie. Étant donné que l'étiquette de réponse correcte elle-même est indirectement utilisée comme quantité de caractéristiques, il est nécessaire de faire attention aux fuites et sa mise en œuvre est assez compliquée. La raison pour laquelle j'avais besoin de cette technique cette fois était parce qu'il y avait une variable catégorielle appelée "place_patient_live2" avec 477 catégories. Normalement, lors de l'utilisation d'une variable catégorielle dans NN, elle est traitée par OneHotEncoding (dans GBDT, la variable catégorielle peut être utilisée telle quelle), mais lors de la création de 477 colonnes et une seule d'entre elles a du sens, beaucoup Vous pouvez voir que la colonne devient un montant de fonctionnalité inutile (= matrice éparse) Par conséquent, il était nécessaire de réduire considérablement le nombre de colonnes (477 → 1) en utilisant le codage cible afin que l'apprentissage puisse être effectué efficacement. En fait, ce dernier était beaucoup plus précis dans mon modèle lorsqu'il était formé avec OneHotEncoding et lorsqu'il était formé avec l'encodage cible.
3-2. Pseudo Labeling Le pseudo-étiquetage est une méthode d'apprentissage semi-supervisée. Nous visons à améliorer la précision en apprenant non seulement les données d'entraînement, mais également les données de test. Tout d'abord, entraînez les données d'entraînement avec un modèle normal, évaluez les données de test et donnez la valeur prédite, mais utilisez cette valeur prédite telle quelle en tant qu'étiquette des données de test, et entraînez-vous à nouveau avec les données d'entraînement + test à partir de 0. Je vais. En conséquence, si le résultat de la prédiction d'apprentissage est correct dans une certaine mesure, la diversité des données de test peut être obtenue. Lors de l'utilisation de cette méthode, il est nécessaire de disposer de données de test suffisantes et elle ne peut pas être utilisée dans des compétitions telles que Code Competition où presque aucune donnée de test n'est fournie. Vous devez également faire attention à la quantité de données de test que vous utilisez, et il est dit qu'il est préférable d'utiliser une quantité telle que train: test = 2: 1.
En utilisant cette méthode, la précision d'un seul modèle peut être déterminée.
- Public LB 0.96696→0.96733 Et obtenu une amélioration significative de la précision.
Cette fois, nous avons utilisé les résultats de sortie du modèle NN et de l'ensemble de modèles LGBM comme étiquettes de données de test.
4. Vérification
4-1. Politique
À l'origine, avant le début de cette compétition, j'avais une idée approximative qu'il s'agirait de données de table, alors allons-y avec le flux d'EDA → Créer une base de référence avec LightGBM → Extraction du poids de la quantité de fonctionnalités → Implémentation NN! Cependant, au début de la compétition, la mise en œuvre par EDA et LightGBM par M. Akiyama, l'opération et le poids de la quantité de fonctionnalités ont été publiés, il n'y avait donc plus grand chose à faire. (J'avais hâte de l'implémenter à partir de zéro, donc c'est un peu décevant pour ceux qui ont connu la concurrence ...)
Cela dit, la précision de LightGBM était à peu près comprise, j'ai donc implémenté le modèle linéaire avec Pytorch comme base de référence. (Pour le moment, la politique était de commencer à créer des quantités de fonctionnalités avec LightGBM tel quel, mais comme je n'ai pas beaucoup de connaissances sur l'ingénierie des fonctionnalités, j'ai soudainement commencé à créer NN.)
4-2. Sélection et utilisation des fonctionnalités à utiliser
En ce qui concerne la quantité de fonctionnalités à utiliser, j'ai utilisé presque tous les modèles LGBM qui ont déjà été publiés. Étant donné que la méthode de création de la quantité de caractéristiques à appliquer est différente pour chacun, celle utilisée à la ligne de base est décrite en gros ci-dessous.
** ・ Standardisation ** Standardisation. Échelle à moyenne 0, variance 1.
standard_cols = [
"age",
"entry_-_symptom_date",
"entry_date_count",
"date_symptoms",
"entry_date",
]
** ・ Encodage Onehot ** Traitement des variables catégorielles. Créez autant de colonnes que de catégories et définissez 1 pour cette colonne et 0 pour les autres.
onehot_cols = [
"place_hospital",
"place_patient_birth",
"type_hospital",
"contact_other_covid",
"test_result",
"pneumonia",
"intubed",
"patient_type",
"chronic_renal_failure",
"diabetes",
"icu",
"obesity",
"immunosuppression",
"sex",
"other_disease",
"pregnancy",
"hypertension",
"cardiovascular",
"asthma",
"tobacco",
"copd",
]
** ・ Encodage cible ** Une méthode qui utilise la moyenne des étiquettes correctes pour toute la catégorie en tant que quantité d'entités target_E_cols = ["place_patient_live2"]
・ Données de date Tout d'abord, changez les données données sous forme de date (2020/09/26) en données pouvant être considérées comme une valeur continue (exemple: 9/27: 1, 9/28: 2 lorsque 9/26 vaut 0). .. Après cela, min_max_encoding est effectué avec la valeur maximale définie sur 1 et la valeur minimale sur 0.
4-3. Référence
name : medcon2020_tachyon_baseline
about : simple NN baseline
model : Liner Model
batch : 32
epoch : 20
criterion : BCEWithLogitsLoss
optimizer : Adam
init_lr : 1e-2
scheduler: CosineAnnealingLR
data : plane
preprocess : OnehotEncoding,Standardization,Target Encoding
train_test_split : StratifiedKFold, k=5
Public LB : 0.96575
4-4. Modèle LightGBM
En ce qui concerne la quantité de caractéristiques, le réglage des paramètres a été effectué en appliquant Optuna en utilisant le résultat d'ingénierie des caractéristiques de M. Akiyama tel quel. résultat,
La ligne de base d'Akiyama: Public LB0.96636
Modèle utilisant Optuna: Public LB0.96635
Puisque presque le même résultat a été obtenu, on peut dire que cette base de référence était un modèle assez complet. Cependant, comme les modèles ont des paramètres différents dans une certaine mesure, l'effet d'ensemble (amélioration de la précision due à la diversité) peut être attendu dans une certaine mesure.
4-5. Soumission d'ensemble
Après avoir ajusté les paramètres de base de 4-2 et obtenu une certaine précision, j'ai d'abord soumis un modèle NN et la moyenne du modèle de base d'Akiyama.
※Public LB
NN seul: 0.96696
LGBM seul: 0.96636
Average Ensembling : 0.96738
C'est une amélioration inattendue de la précision. Les algorithmes de NN et GBDT étant complètement différents, on considère que le score a augmenté de manière significative. A cette époque, j'étais classé numéro un à Butchigiri.
4-6.Pseudo Labeling Le deuxième jour (dernier jour), nous avons implémenté le pseudo étiquetage expliqué dans 3. Théorie. Il y avait peut-être un moyen plus rapide d'améliorer la précision, mais c'est le seul qui m'est venu à l'esprit ...
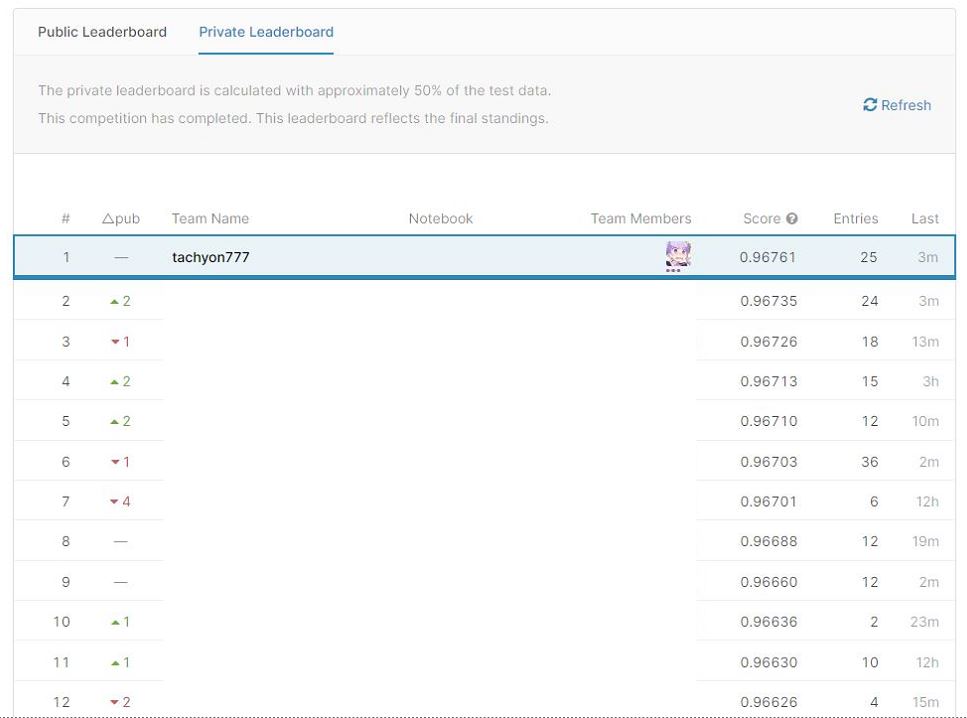
Avant d'implémenter le pseudo-étiquetage Score d'ensemblage moyen: privé: 0,96753 public: 0,96761 Après la mise en œuvre du pseudo-étiquetage Score d'ensemblage moyen: ** Privé: 0,96761 ** Public: 0,96768
En fin de compte, j'ai choisi ces deux éléments comme soumission finale. En conséquence, ce dernier est devenu le modèle gagnant.
4-7.Pipeline
Excusez-moi pour la diapositive PowerPoint, mais je l'ai finalement terminée avec le pipeline suivant.
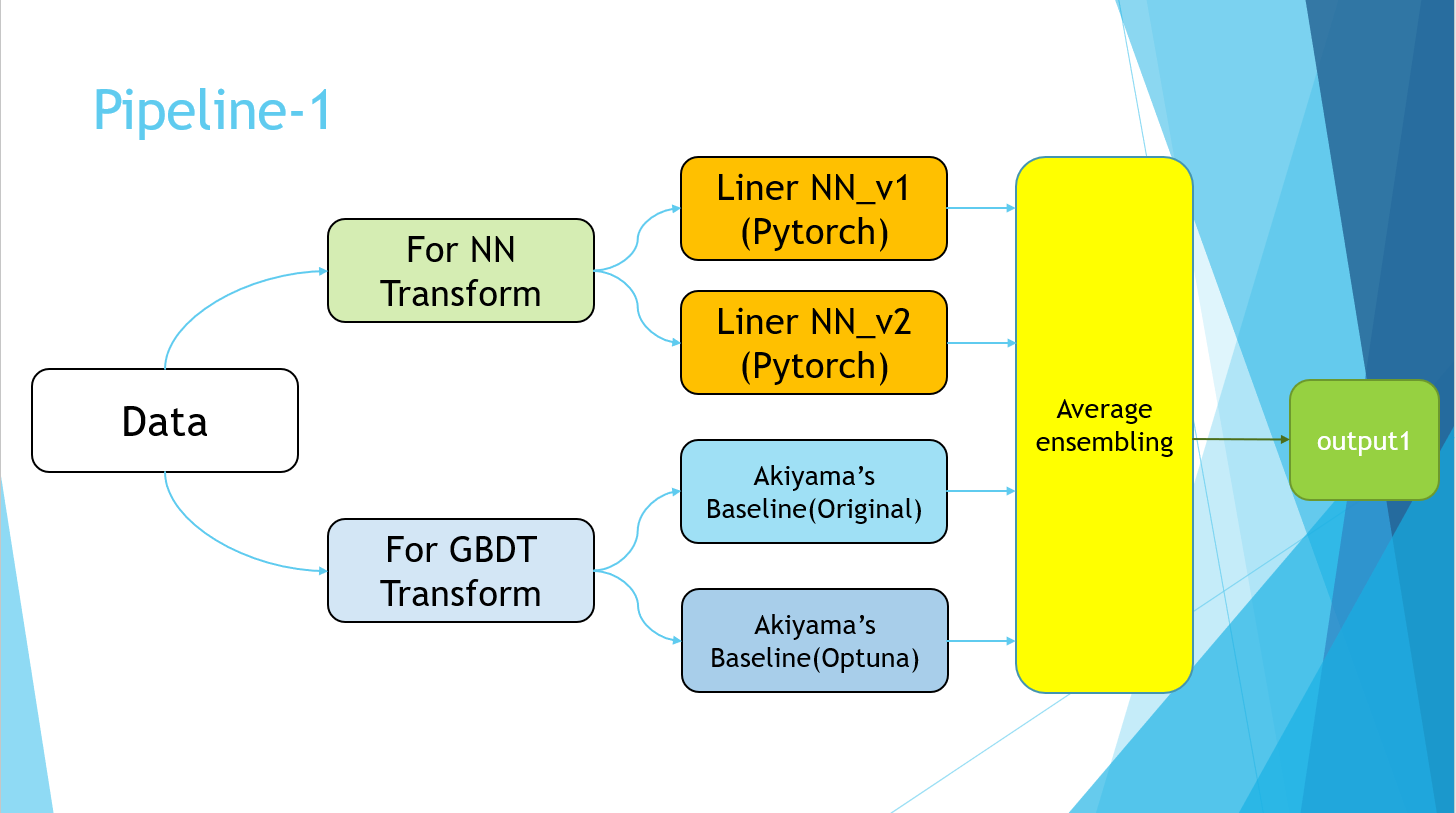
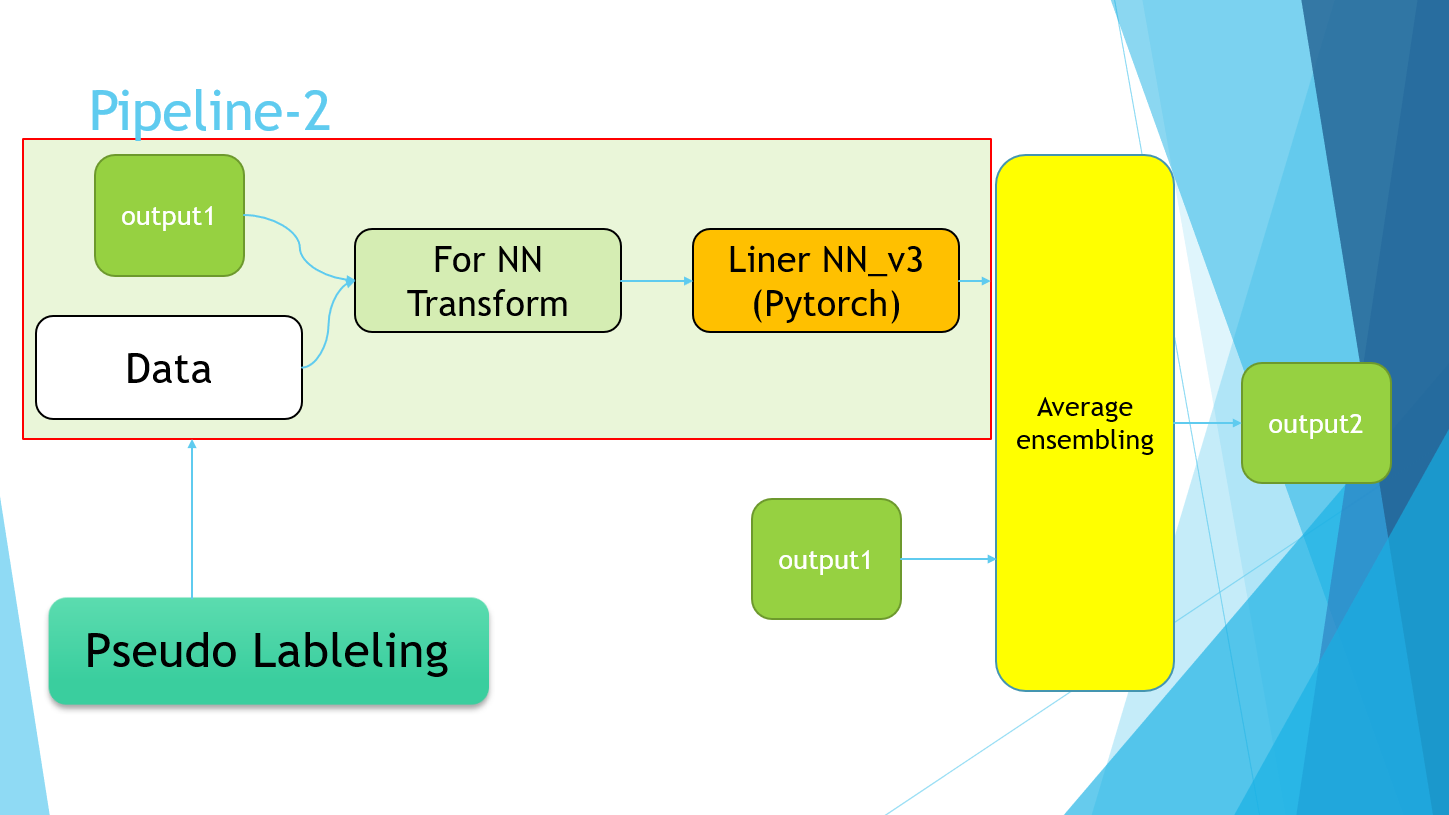
La partie Pipeline-2 fait simplement référence à l'implémentation du pseudo-étiquetage. Enfin, les valeurs moyennes des quatre modèles de Pipeline-1 et des modèles entraînés à l'aide des données de pseudo-étiquetage de Pipeline-2 ont été prises.
5. Résultat
A gagné. (Avez-vous déjà remporté le championnat de votre vie ...?) Quant à la méthode, je sens que je l'ai parfaitement fait et j'ai un sentiment d'accomplissement. Le deuxième jour, j'étais tout le temps à la première place et j'avais peur de ne pas être dépassé, donc j'étais plus soulagé que content ...
6. Examen
Puisqu'il s'agit d'un concours privé, je m'abstiendrai de soulever la solution d'autres personnes. Une autre chose que je voulais essayer, c'est du haut ・ Création de la quantité de fonctionnalités ・ Boost de chat ・ Hyper réglage des paramètres du modèle NN C'est comme ça. Si une compétition était réellement organisée avec ces données sur le kaggle, mon score serait inférieur à la médaille de bronze, donc je pense qu'il y a encore place à l'amélioration.
7. À la fin
Merci à tous les organisateurs, y compris l'AI & Machine Learning Society (AIMS) de l'Université d'Osaka, aux présentateurs du premier jour et aux participants au concours! J'espère que ma solution sera utile autant que possible.
8. Références
- Daisuke Kadowaki Takashi Sakata Keisuke Hosaka Yuji Hiramatsu Technologie d'analyse de données gagnée avec Kaggle Technology Review Company 2019
Recommended Posts