Entfernen Sie DICOM-Bilder in Python
Einfach ausgedrückt ist ein DICOM-Bild ein Bild, das im Kommunikationsprotokoll (DICOM) zum Austausch medizinischer Bilder zwischen Geräten verarbeitet wird, und dem Bild werden verschiedene zusätzliche Informationen (Patienteninformationen, Patienteninformationen usw.) hinzugefügt. Dies ist der Gerätetyp, die Informationen des Bildes selbst und verschiedene Metadaten.
Wenn ein Arzt eine Untersuchung wie CT oder MRT durchführt, werden Sie möglicherweise von einigen Krankenhäusern aufgefordert, das Bild als Daten zu verwenden. Wenn das Format zu diesem Zeitpunkt die Erweiterung .dcm ist, handelt es sich um ein DICOM-Image.
Vorbereiten der Arbeit mit DICOM in Python
Wenn Sie mit DICOM-Images in Python arbeiten möchten, werden Sie anscheinend pydicom verwenden.
% anaconda show conda-forge/pydicom
Using Anaconda API: https://api.anaconda.org
Name: pydicom
Summary: Pure python package for DICOM medical file reading and writing
Access: public
Package Types: conda
Versions:
+ 0.9.9
To install this package with conda run:
conda install --channel https://conda.anaconda.org/conda-forge pydicom
Deshalb `conda install -c https://conda.anaconda.org/conda-forge pydicom `.
Bereiten Sie ein Beispiel für ein DICOM-Bild vor
Wenn Sie leider kein eigenes MRT-Bild haben, laden Sie ein Beispielbild herunter. Auf der Osirix-Website, die als DICOM-Viewer bekannt ist, befindet sich DICOM Image Library. Vorerst habe ich ein MRT-Bild eines Gehirntumors namens [BRAINIX] heruntergeladen (http://www.osirix-viewer.com/datasets/DATA/BRAINIX.zip). Wenn Sie diese ZIP-Datei nach der Bildgebung mit MRT erweitern, gibt es Bilder (T1-Betonung, so ein Typ), die auf verschiedene Weise verarbeitet wurden, damit der Dolmetscher sie sehen kann, aber in dieser "T1-3D-FFE-C" Ich habe beschlossen, 100 Dateien im Ordner "\ - \ 801 /" zu verwenden.
Lesen Sie das DICOM-Bild
Unten finden Sie ein Beispiel zum Laden eines Bildes mit der Funktion pydicom.
Lesen Sie ein DICOM-Bild und beziehen Sie sich auf die Metadaten
Laden Sie das Bild und zeigen Sie den DICOM-Header an.
import dicom
d = dicom.read_file('BRAINIX/IM-0001-0075.dcm')
print(d)
Übrigens, wenn Sie bestimmte Metadaten anzeigen möchten, gehen Sie wie folgt vor.
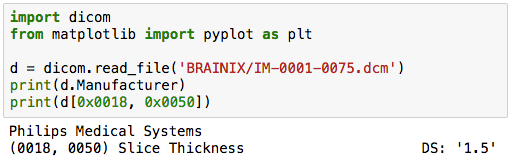
print(d.Manufacturer)
Anscheinend ist der Gerätehersteller "Philips Medical Systems".
print(d[0x0018, 0x0050])
Anscheinend beträgt die Scheibendicke 1,5 mm.
Auf diese Weise haben die DICOM-Metadaten einen Schlüsselnamen, der als Tag bezeichnet wird, und die Schlüssel sind nummeriert. Und mit pydicom können Sie entweder den Schlüsselnamen oder die Nummer anzeigen.
Lesen Sie ein DICOM-Bild und zeigen Sie das Bild an
Da der eigentliche Bildteil des DICOM-Bildes im Array-Format numpy extrahiert werden kann, kann er entweder mit opencv oder matplotlib angezeigt werden.

Liest mehrere DICOM-Bilder und konvertiert sie in ein kontinuierliches tomografisches Bild
Das diesmal verwendete BRAINIX-Bild besteht übrigens aus einer Serie von 100 Bildern mit einer Dicke von 1,5 mm. Diese 100 Bilder können gelesen und in ein 3D-Array importiert werden.
import dicom
from matplotlib import pyplot as plt
%matplotlib inline
root_dir = './BRAINIX'
dcms = []
for d, s, fl in os.walk(root_dir):
for fn in fl:
if ".dcm" in fn.lower():
dcms.append(os.path.join(d, fn))
ref_dicom = dicom.read_file(dcms[0])
d_array = np.zeros((ref_dicom.Rows, ref_dicom.Columns, len(dcms)), dtype=ref_dicom.pixel_array.dtype)
for dcm in dcms:
d = dicom.read_file(dcm)
d_array[:, :, dcms.index(dcm)] = d.pixel_array
Kurz gesagt, alle Dateien im BRAINIX-Ordner mit der Erweiterung .dcm werden gelesen und der Inhalt wird in ein dreidimensionales Array namens d_array kopiert.
Wenn Sie ein Slice anzeigen möchten, beispielsweise das 50. Slice, führen Sie einfach `pyplt.imshow (d_array [:,:, 49])` aus.
Nachdem es in ein dreidimensionales Array eingelesen wurde, können drei Abschnitte angezeigt werden.
import os
import dicom
from matplotlib import pyplot as plt
%matplotlib inline
#Lesen Sie die dcm-Datei im BRAINIX-Ordner
root_dir = './BRAINIX'
dcms = []
for d, s, fl in os.walk(root_dir):
for fn in fl:
if ".dcm" in fn.lower():
dcms.append(os.path.join(d, fn))
ref_dicom = dicom.read_file(dcms[0])
d_array = np.zeros((ref_dicom.Rows, ref_dicom.Columns, len(dcms)), dtype=ref_dicom.pixel_array.dtype)
for dcm in dcms:
d = dicom.read_file(dcm)
d_array[:, :, dcms.index(dcm)] = d.pixel_array
#3 Querschnittsanzeige
print(d_array.shape)
plt.subplot(1, 3, 1)
plt.imshow(d_array[127, :, :])
plt.subplot(1, 3, 2)
plt.imshow(d_array[:, 127, :])
plt.subplot(1, 3, 3)
plt.imshow(d_array[:, :, 49])

Verarbeiten und speichern Sie DICOM-Bilder
Wenn Sie das gelesene DICOM-Bild verarbeiten und durch Überschreiben des ursprünglichen DICOM-Bildes speichern möchten, müssen Sie das Array von pixel_array bearbeiten.
Das Originalbild ändert sich jedoch nicht nur durch Bearbeiten des Arrays von pixel_array. Dies liegt daran, dass die tatsächlichen Bilddaten des DICOM-Bildes in einem Array namens PixelData gespeichert sind und die Schnittstelle zum Lesen pixel_array ist. Wenn der Inhalt des bearbeiteten pixel_array beispielsweise in einem Array namens img gespeichert wird, muss der Inhalt daher in PixelData zurückgeschrieben werden.
d = dicom.read_file(dcm)
img = d.pixel_array
#Verarbeitung von img
d.PixelData = img.tostring()
d.save_as(dcm)
Versuchen Sie, das DICOM-Bild in einem Kreis zuzuschneiden
Ich weiß nicht, was es ist, aber ich werde versuchen, "das DICOM-Bild nur im Mittelteil mit einem Kreis mit einem Durchmesser auszuschneiden, der halb so lang und breit wie das Bild ist".
Der folgende Code erstellt ein Bild mit 1 auf der Innenseite und 0 auf der Außenseite eines Kreises mit einem Durchmesser, der halb so lang und breit wie das Bild ist. Wenn der Radius r ist, kann die vertikale und horizontale Größe des Bildes aus ref_dicom.pixel_array.shape abgerufen werden. Stellen Sie also 1/4 dieser Größe ein.
r = ref_dicom.pixel_array.shape[0] / 4
x, y = np.indices((ref_dicom.Rows, ref_dicom.Columns))
circle = (x - (ref_dicom.Columns / 2))**2 + (y - (ref_dicom.Rows / 2))**2 < r**2
mask = circle.astype(int)
Durch Multiplizieren dieser Maske ist die Innenseite des Kreises 1 und die Außenseite 0, sodass sie in einem Kreis ausgeschnitten werden kann.
###Lesen Sie mehrere DICOM-Bilddateien und schneiden Sie den Mittelteil in einem Kreis aus
import os
import dicom
from matplotlib import pyplot as plt
%matplotlib inline
#Lesen Sie die dcm-Datei im BRAINIX-Ordner
root_dir = './BRAINIX'
dcms = []
for d, s, fl in os.walk(root_dir):
for fn in fl:
if ".dcm" in fn.lower():
dcms.append(os.path.join(d, fn))
ref_dicom = dicom.read_file(dcms[0])
#Erstellen Sie eine Maske, die in einen Kreis ausgeschnitten werden soll
r = ref_dicom.pixel_array.shape[0] / 4
x, y = np.indices((ref_dicom.Rows, ref_dicom.Columns))
circle = (x - (ref_dicom.Columns / 2))**2 + (y - (ref_dicom.Rows / 2))**2 < r**2
mask = circle.astype(int)
#Führen Sie eine Maskenverarbeitung für alle Bilder durch
d_array = np.zeros((ref_dicom.Rows, ref_dicom.Columns, len(dcms)), dtype=ref_dicom.pixel_array.dtype)
for dcm in dcms:
d = dicom.read_file(dcm)
img = d.pixel_array * mask
d_array[:, :, dcms.index(dcm)] = img
#3 Querschnittsanzeige
plt.figure(figsize=(8, 6))
plt.subplot(1, 3, 1)
plt.imshow(d_array[127, :, :])
plt.subplot(1, 3, 2)
plt.imshow(d_array[:, 127, :])
plt.subplot(1, 3, 3)
plt.imshow(d_array[:, :, 49])
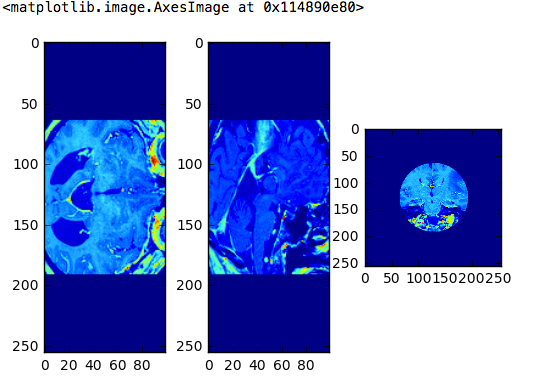
Beim Umgang mit DICOM-Bildern in Python sieht es so aus.
Der heutige Code
- https://github.com/fukuit/Python_SelfLearning/tree/master/dicom
Recommended Posts