[PYTHON] [Veraltet] Chainer v1.24.0 Anfänger-Tutorial
Warnung
Dieser Artikel ist zu alt, um lächerlich zu sein. Er wurde für die endgültige Veröffentlichung von Chainer v1 (v1.24.0) geschrieben, die nicht mehr unterstützt wird.
** Es gibt einen Artikel für die neueste stabile Version von Chainer v3 ab Dezember 2017 unter hier, daher ist es unbedingt erforderlich, mit v1 zu beginnen. Wenn Sie keine besondere Situation haben, fliegen Sie jetzt zu dort. ** ** **
Los !! -> Chainer v3 Anfänger-Tutorial
1st Chainer Beginner's Hands-on fand im Mehrzweckraum des Preferred Networks-Büros in Otemachi statt. Dieser Artikel ist ein Artikel über das, was ich in diesem praktischen Artikel gemacht habe.
Die am Tag der praktischen Anwendung verwendeten Materialien sind im folgenden Github-Repository zusammengefasst.
Am Tag der Veranstaltung wurden 20 GPU-Server mit 4 Pascal TITAN X (insgesamt 80 GPUs!) Kostenlos von Sakura Internet ausgeliehen und zum Anfassen abgehalten, damit alle Teilnehmer sie nutzen konnten. Wir möchten diese Gelegenheit nutzen, um Sakura Internet zu danken. Es wird gesagt, dass Sakura High Thermal Power Computing in Kürze stündlich einen GPU-Server mieten wird. Wenn Sie also eine GPU-Umgebung einführen möchten, lesen Sie diese bitte durch.
Sakura High Thermal Power Computing
Am Tag der praktischen Arbeit habe ich mich zunächst bei ssh bei jedem Knoten dieser geliehenen Sakura-Hochwärmeleistung angemeldet und NVIDIA CUDA installiert. In diesem Artikel habe ich diesen Teil übersprungen und aus dem Teil über die Verwendung von Chainer zusammengefasst Ich werde fortsetzen.
Informationen zum Aufbau der Umgebung finden Sie in den folgenden Materialien.
Umgebungskonstruktion auf dem Sakura High-Heat-Computing-Server
Dies kann mit einigen Ausnahmen als Verfahren zum Erstellen einer Umgebung für einen Server verwendet werden, der mit einer NVIDIA-GPU ausgestattet ist, die unter Ubuntu 14.04 ausgeführt wird.
Kommen wir nun zum Hauptthema. Das folgende Tutorial basiert auf Python 3.4, das standardmäßig unter Ubuntu 14.04 installiert ist. Bitte lesen Sie vorab die Seiten 9 und 11 des obigen Dokuments, um die zugehörigen Bibliotheken und den Chainer selbst zu installieren. ** Es wird davon ausgegangen, dass der folgende Codeteil und das folgende Ausgabeergebnis auf dem Jupyter-Notebook ausgeführt werden. ** ** **
Schreiben wir eine Lernschleife
Hier,
- Extrahieren Sie Daten aus dem Datensatz
- Füllen Sie das Modell aus
- Verwenden Sie das Optimierungsprogramm, um die Modellparameter zu aktualisieren und eine Trainingsschleife auszuführen
Ich werde das versuchen. Auf diese Weise können Sie lernen, wie Sie eine Lernschleife schreiben, ohne Trainer zu verwenden.
1. Datensatzvorbereitung
Hier verwenden wir eine bequeme Methode zur Verwendung des von Chainer bereitgestellten MNIST-Datensatzes. Damit ist alles verborgen, vom Herunterladen von Daten bis zum Abrufen einzelner Daten.
from chainer.datasets import mnist
#Wenn der Datensatz nicht heruntergeladen wurde, laden Sie ihn ebenfalls herunter
train, test = mnist.get_mnist(withlabel=True, ndim=1)
#Erstellt das Ergebnis der Grafikzeichnung mithilfe der im Notizbuch angezeigten Matplotlib.
%matplotlib inline
import matplotlib.pyplot as plt
#Datenbeispiel
x, t = train[0]
plt.imshow(x.reshape(28, 28), cmap='gray')
plt.show()
print('label:', t)
Ausgabeergebnis:
Downloading from http://yann.lecun.com/exdb/mnist/train-images-idx3-ubyte.gz...
Downloading from http://yann.lecun.com/exdb/mnist/train-labels-idx1-ubyte.gz...
Downloading from http://yann.lecun.com/exdb/mnist/t10k-images-idx3-ubyte.gz...
Downloading from http://yann.lecun.com/exdb/mnist/t10k-labels-idx1-ubyte.gz...
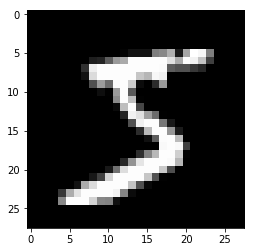
label: 5
2. Erstellen Sie einen Iterator
Erstellen wir einen "Iterator", der eine feste Anzahl von Daten aus dem Datensatz abruft und diese bündelt, um einen Mini-Batch zu erstellen und diesen zurückzugeben. Wir werden dies in der folgenden Lernschleife verwenden. Der Iterator gibt einen neuen Mini-Batch mit der Methode next () zurück. Intern verfügt es über Eigenschaften (is_new_epoch), die steuern, wie viele Runden der Datensatz geleckt wurde ( epoch) und ob die aktuelle Iteration die erste Iteration einer neuen Epoche ist.
from chainer import iterators
batchsize = 128
train_iter = iterators.SerialIterator(train, batchsize)
test_iter = iterators.SerialIterator(test, batchsize,
repeat=False, shuffle=False)
Über Iterator
--SerialIterator`, eine Art von Iterator, der von Chainer erstellt wurde, ist der einfachste Iterator, der die Daten im Datensatz der Reihe nach abruft.
- Nehmen Sie das Dataset-Objekt und die Stapelgröße als Argumente.
- Wenn Sie für viele Runden wiederholt Daten aus dem zu diesem Zeitpunkt übergebenen Datensatzobjekt lesen müssen, setzen Sie das Argument "repeat" auf "True". Wenn Sie nach einer Runde keine weiteren Daten mehr abrufen möchten, ist dies der Fall Sei "falsch". Der Standardwert ist "True".
- Wenn Sie "True" an das Argument "Shuffle" übergeben, wird die Reihenfolge der aus dem Datensatz abgerufenen Daten für jede Epoche zufällig geändert.
Da hier "batchsize = 128" eingestellt ist, sind der "train_iter", der der "Iterator" für die Trainingsdaten ist, und der "test_iter", der der "Iterator" für die hier erzeugten Testdaten ist, 128 numerische Bilddaten. Wird "Iterator" genannt, der auf einmal zurückgibt. [^ Trainingsdaten und Validierungsdaten]
3. Modelldefinition
Hier definieren wir ein einfaches dreischichtiges Perzeptron. Dies ist ein Netzwerk, das nur aus vollständig verbundenen Schichten besteht. Die Anzahl der Einheiten in der mittleren Ebene ist entsprechend auf 100 eingestellt, und die Ausgabe beträgt 10 Klassen. Setzen Sie sie daher auf 10. Dies liegt daran, dass der hier verwendete MNIST-Datensatz 10 verschiedene Bezeichnungen hat. Lassen Sie uns nun einen kurzen Blick auf die zum Definieren des Modells erforderlichen "Links", "Funktionen" und "Ketten" werfen.
Link und Funktion
--Chainer unterscheidet jede Schicht des neuronalen Netzwerks in "Link" und "Funktion".
- **
Linkist eine Funktion mit Parametern. ** ** ** - **
Funktionist eine Funktion ohne Parameter. ** ** ** - Kombinieren Sie diese, um das Modell zu beschreiben.
- Unter dem Modul
chainer.linksbefinden sich viele Ebenen mit Parametern. - Unter dem Modul "chainer.functions" befinden sich viele Ebenen ohne Parameter.
- Um diese einfach zu bedienen zu machen
import chainer.links as L
import chainer.functions as F
Es ist üblich, ihm einen Alias zu geben und ihn wie "L.Convolution2D (...)" oder "F.relu (...)" zu verwenden.
Chain
--Chain ist eine Klasse ** zum Gruppieren von Ebenen mit Parametern = ** Link.
- Parameter zu haben bedeutet im Grunde, dass Sie sie beim Trainieren des Modells aktualisieren müssen (mit Ausnahmen).
- Um alle Parameter zu erhalten, die der Optimierer während des Lernens aktualisieren sollte, werden wir sie mit Chain an einer Stelle zusammenfügen.
Ein Modell, das durch das Erben von Chain definiert wird
- Das Modell wird häufig als eine Klasse definiert, die von der "Chain" -Klasse erbt.
- In diesem Fall wird im Konstruktor der Klasse, die das Modell darstellt, der Name der zu registrierenden Ebene in Form eines Schlüsselwortarguments und des Objekts an den Konstruktor der übergeordneten Klasse automatisch in der Form beibehalten, die im Optimierer gefunden wurde. Werde es verlassen.
- Dies kann auch an anderer Stelle mit der Methode "add_link" erfolgen.
- Außerdem ist es praktisch, die Methode call zu definieren und den darin enthaltenen Weiterleitungsprozess zu beschreiben, damit Sie Daten mit dem Accessor
()wie einen Funktionsaufruf an das Modell übergeben können.
Auf GPU ausführen
- Die
Chain-Klasse hat eineto_gpu-Methode. Wenn Sie in diesem Argument eine GPU-ID angeben, werden alle Parameter des Modells in den Speicher der angegebenen GPU-ID übertragen. - Dies ist erforderlich, um Vorwärts- / Rückwärtsberechnungen innerhalb des Modells auf dieser angegebenen GPU durchzuführen.
- Wenn Sie dies nicht tun, werden diese Dinge auf der CPU erledigt.
Definieren wir nun das Modell. Lassen Sie uns zunächst den Zufallszahlen-Startwert korrigieren, damit wir fast das gleiche Ergebnis wie in diesem Artikel reproduzieren können. (Wenn Sie die Reproduzierbarkeit des Berechnungsergebnisses strenger gewährleisten möchten, müssen Sie die Option "deterministisch" kennen. Dieser Artikel ist nützlich: Warum und die Gegenmaßnahme, dass sich das Ergebnis jedes Mal ändert, wenn die GPU in Chainer verwendet wird //qiita.com/TokyoMickey/items/cc8cd43545f2656b1cbd).
import numpy
numpy.random.seed(0)
import chainer
if chainer.cuda.available:
chainer.cuda.cupy.random.seed(0)
Definieren wir nun das Modell, erstellen ein Objekt und senden es an die GPU.
import chainer
import chainer.links as L
import chainer.functions as F
class MLP(chainer.Chain):
def __init__(self, n_mid_units=100, n_out=10):
#Registrierung von Ebenen mit Parametern
super(MLP, self).__init__(
l1=L.Linear(None, n_mid_units),
l2=L.Linear(n_mid_units, n_mid_units),
l3=L.Linear(n_mid_units, n_out),
)
def __call__(self, x):
#Vorwärtsberechnung schreiben, wenn Daten empfangen werden
h1 = F.relu(self.l1(x))
h2 = F.relu(self.l2(h1))
return self.l3(h2)
gpu_id = 0
model = MLP()
model.to_gpu(gpu_id) #Wenn Sie möchten, dass die CPU die Arbeit erledigt, kommentieren Sie diese Zeile aus.
NOTE
Hier bedeutet die Klasse "L.Linear" eine vollständig verbundene Schicht. Wenn Sie zur Laufzeit "None" als erstes Argument des Konstruktors übergeben, werden zur Laufzeit Daten in diese Ebene eingegeben, die erforderliche Anzahl von Eingabeeinheiten wird automatisch berechnet und "(n_input)" $ \ times $ Erstellen Sie eine Matrix der Größe "n_mid_units" und behalten Sie sie als Parameter bei. Dies ist später eine nützliche Funktion, beispielsweise wenn die Faltungsschicht vor der vollständig verbundenen Schicht platziert wird.
Wie bereits erwähnt, verfügt "Link" über einen Parameter, mit dem Sie auf den Wert dieses Parameters zugreifen können. Zum Beispiel hat das Modell "MLP" oben eine vollständig verbundene Schicht mit dem Namen "l1" registriert. Diese vollständig gekoppelte Phase hat zwei Parameter, "W" und "b". Diese sind von außen zugänglich. Um beispielsweise auf b zuzugreifen, gehen Sie wie folgt vor:
print('Die Form des Vorspannungsparameters der ersten vollständig gekoppelten Phase ist', model.l1.b.shape)
print('Der Wert unmittelbar nach der Initialisierung ist', model.l1.b.data)
Ausgabeergebnis
Die Form des Vorspannungsparameters der ersten vollständig gekoppelten Phase ist(100,)
Der Wert unmittelbar nach der Initialisierung ist[ 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0.]
Wenn ich jetzt versuche, auf model.l1.W zuzugreifen, wird folgende Fehlermeldung angezeigt:
AttributeError: 'Linear' object has no attribute 'W'
Dies liegt daran, dass in der Definition des obigen Modells "Keine" als erstes Argument des Konstruktors der "linearen" Verknüpfung übergeben wird, sodass die Matrix "W" erst zum Zeitpunkt der Ausführung zugewiesen wird. Es existiert nicht, aber es ist innerhalb des "linearen" Objekts bekannt, dass es existieren wird.
4. Auswahl der Optimierungsmethode
Chainer bietet viele Optimierungstechniken. Sie befinden sich unter dem Modul "chainer.optimizers". Hier verwenden wir optimizers.SGD, die einfachste Methode zum Gradientenabstieg. Übergeben Sie das Modell (Chain-Objekt) mit der setup-Methode an das Optimizer-Objekt. Dadurch kann das Optimierungsprogramm automatisch den Parametern im Modell folgen, die aktualisiert werden sollen.
Sie können ganz einfach verschiedene andere Optimierungsmethoden ausprobieren. Probieren Sie sie aus und sehen Sie, wie sich die Ergebnisse ändern. Ändern Sie beispielsweise in der folgenden Datei "chainer.optimizers.SGD" den Teil "SGD" in "Momentum SGD", "RMSprop", "Adam" usw. und sehen Sie den Unterschied in den Ergebnissen.
from chainer import optimizers
optimizer = optimizers.SGD(lr=0.01)
optimizer.setup(model)
NOTE
Diesmal habe ich dem Argument lr im SGD-Konstruktor $ 0.01 $ gegeben. Dieser Wert wird als Lernrate bezeichnet und ist als wichtiger ** Hyperparameter ** bekannt, der angepasst werden muss, um das Modell für eine gute Leistung gut zu trainieren.
5. Lernschleife
Es ist endlich eine Lernschleife. Da dies ein Klassifizierungsproblem ist, verwenden wir die Verlustfunktion "softmax_cross_entropy", um den Wert des Verlusts zu berechnen, der minimiert werden soll.
In Chainer wird die Vorwärtsberechnung des Modells unter Verwendung von "Funktion" und "Verknüpfung" durchgeführt. Das Ergebnis und das richtige Antwortetikett werden an die Verlustfunktion übergeben, die eine Art "Funktion" ist und den Skalarwert zurückgibt. Wenn der Verlust berechnet wird, ist dies der Fall Wie jedes andere "Link" oder "Funktion" gibt es ein "Variablen" -Objekt zurück. Da das Objekt "Variable" eine Referenz enthält, um in umgekehrter Richtung zum vorherigen Berechnungsprozess zurückzukehren, rufen Sie einfach die Methode "Variable.backward ()" auf, und die Berechnung von dort aus wird automatisch durchgeführt. Es geht zurück in den Prozess und berechnet den Gradienten der Parameter, die für die in der Mitte durchgeführte Berechnung verwendet wurden.
Mit anderen Worten, die folgenden vier Elemente werden in einer Lernschleife ausgeführt.
- Übergeben Sie die Daten an das Modell und erhalten Sie die Ausgabe "y"
- Berechnen Sie mit
yund der richtigen Bezeichnungtden zu minimierenden Verlustwert mit der Funktionsoftmax_cross_entropy. - Rufen Sie die Rückwärtsmethode der Ausgabevariablen der Funktion softmax_cross_entropy auf, um den Parametern im Modell die Eigenschaft grad zu geben (dies ist der Gradient, der zum Aktualisieren der Parameter verwendet wird).
- Rufen Sie die "update" -Methode des Optimierers auf und aktualisieren Sie alle Parameter mit dem in 3 berechneten "grad".
das ist alles. Wenn Sie an einem einfachen Regressionsproblem anstelle eines Klassifizierungsproblems arbeiten, können Sie "F.mean_squared_error" anstelle von "F.softmax_cross_entropy" verwenden. Darüber hinaus bietet Chainer verschiedene Verlustfunktionen, um verschiedene Problemeinstellungen zu behandeln. Sie können die Liste hier sehen: Verlustfunktionen.
Schreiben wir nun eine Trainingsschleife.
import numpy as np
from chainer.dataset import concat_examples
from chainer.cuda import to_cpu
max_epoch = 10
while train_iter.epoch < max_epoch:
# ----------1 Iteration des Lernens----------
train_batch = train_iter.next()
x, t = concat_examples(train_batch, gpu_id)
#Berechnung des vorhergesagten Wertes
y = model(x)
#Verlustberechnung
loss = F.softmax_cross_entropy(y, t)
#Gradientenberechnung
model.cleargrads()
loss.backward()
#Parameteraktualisierung
optimizer.update()
# ---------------Bisher----------------
#Messen Sie die Vorhersagegenauigkeit für Validierungsdaten am Ende jeder Epoche.
#Lassen Sie uns überprüfen, ob die Generalisierungsleistung des Modells verbessert ist
if train_iter.is_new_epoch: #Wenn 1 Epoche vorbei ist
#Anzeige des Verlustes
print('epoch:{:02d} train_loss:{:.04f} '.format(
train_iter.epoch, float(to_cpu(loss.data))), end='')
test_losses = []
test_accuracies = []
while True:
test_batch = test_iter.next()
x_test, t_test = concat_examples(test_batch, gpu_id)
#Daten weiterleiten
y_test = model(x_test)
#Verlust berechnen
loss_test = F.softmax_cross_entropy(y_test, t_test)
test_losses.append(to_cpu(loss_test.data))
#Berechnen Sie die Genauigkeit
accuracy = F.accuracy(y_test, t_test)
accuracy.to_cpu()
test_accuracies.append(accuracy.data)
if test_iter.is_new_epoch:
test_iter.epoch = 0
test_iter.current_position = 0
test_iter.is_new_epoch = False
test_iter._pushed_position = None
break
print('val_loss:{:.04f} val_accuracy:{:.04f}'.format(
np.mean(test_losses), np.mean(test_accuracies)))
Ausgabeergebnis
epoch:01 train_loss:0.7828 val_loss:0.8276 val_accuracy:0.8167
epoch:02 train_loss:0.3672 val_loss:0.4564 val_accuracy:0.8826
epoch:03 train_loss:0.3069 val_loss:0.3702 val_accuracy:0.8976
epoch:04 train_loss:0.3333 val_loss:0.3307 val_accuracy:0.9078
epoch:05 train_loss:0.3308 val_loss:0.3079 val_accuracy:0.9129
epoch:06 train_loss:0.3210 val_loss:0.2909 val_accuracy:0.9162
epoch:07 train_loss:0.2977 val_loss:0.2781 val_accuracy:0.9213
epoch:08 train_loss:0.2760 val_loss:0.2693 val_accuracy:0.9232
epoch:09 train_loss:0.1762 val_loss:0.2566 val_accuracy:0.9263
epoch:10 train_loss:0.2444 val_loss:0.2479 val_accuracy:0.9284
Die letzten 10 Epochen konzentrieren sich auf "val_accuracy" und betragen $ 0,9286 $. Handschriftliche Zahlen können jetzt mit einer Genauigkeit von ca. 93% klassifiziert werden.
6. Speichern Sie das trainierte Modell
Chainer verfügt über zwei Serialisierungsfunktionen. Zum einen wird das Modell im HDF5-Format und zum anderen im NPZ-Format von NumPy gespeichert. Dieses Mal speichern wir anstelle von HDF5, für das zusätzliche Bibliotheken installiert werden müssen, das Modell im NPZ-Format mithilfe der Serialisierungsfunktion, die von der NumPy-Standardfunktion bereitgestellt wird.
from chainer import serializers
serializers.save_npz('my_mnist.model', model)
#Stellen Sie sicher, dass es richtig gespeichert ist
%ls -la my_mnist.model
\ * Die letzte Zeile funktioniert nur auf dem Jupyter-Notebook.
Ausgabeergebnis
-rw-rw-r-- 1 ubuntu ubuntu 333853 Mar 29 16:51 my_mnist.model
7. Laden Sie das gespeicherte Modell und schließen Sie es ab
Laden Sie die soeben gespeicherte NPZ-Datei und lassen Sie das Netzwerk die Bezeichnung für die Testdaten vorhersagen. Da die Parameter in der NPZ-Datei gespeichert sind, erstellen Sie zunächst ein Objekt des Modells mit der Logik der Vorwärtsberechnung und überschreiben Sie die Parameter mit dem Wert der zuvor gespeicherten NPZ, um den Status des Modells unmittelbar nach dem Training wiederherzustellen. Ich werde.
#Erstellen Sie zunächst ein Objekt desselben Modells
infer_model = MLP()
#Laden Sie gespeicherte Parameter in dieses Objekt
serializers.load_npz('my_mnist.model', infer_model)
#Senden Sie das Modell zur Berechnung auf der GPU an die GPU
infer_model.to_gpu(gpu_id)
#Testdaten
x, t = test[0]
plt.imshow(x.reshape(28, 28), cmap='gray')
plt.show()
print('label:', t)
Ausgabeergebnis

label: 7
Ich habe versucht, die Testdaten anzuzeigen, auf die das Modell von nun an schließen wird. Das Folgende ist ein Beispiel für eine Schlussfolgerung für dieses Bild.
from chainer.cuda import to_gpu
#Machen Sie es in Form einer Mini-Charge (hier ist es eine Mini-Charge der Größe 1,
#Es ist auch möglich, mehrere Mini-Chargen der Größe n) gemeinsam abzuleiten.
print(x.shape, end=' -> ')
x = x[None, ...]
print(x.shape)
#Daten werden auch auf der GPU zur Berechnung auf der GPU gesendet
x = to_gpu(x, 0) #Wenn Sie dies auf der CPU tun möchten, kommentieren Sie hier.
#Übergeben Sie die Vorwärtsfunktion des Modells
y = infer_model(x)
#Nehmen Sie den Inhalt heraus, da er im variablen Format ausgegeben wird
y = y.data
#Senden Sie das Ergebnis an die CPU
y = to_cpu(y) #Wenn Sie dies auf der CPU tun möchten, kommentieren Sie hier.
#Maximaler Index anzeigen
pred_label = y.argmax(axis=1)
print('predicted label:', pred_label[0])
Ausgabeergebnis
(784,) -> (1, 784)
predicted label: 7
Verwenden wir Trainer
Der Trainer macht das explizite Schreiben von Lernschleifen überflüssig. Darüber hinaus erleichtern verschiedene praktische Erweiterungen die Visualisierung und Speicherung von Protokollen.
1. Datensatzvorbereitung
from chainer.datasets import mnist
train, test = mnist.get_mnist()
2. Vorbereitung des Iterators
from chainer import iterators
batchsize = 128
train_iter = iterators.SerialIterator(train, batchsize)
test_iter = iterators.SerialIterator(test, batchsize, False, False)
3. Modellvorbereitung
Hier verwenden wir das gleiche Modell wie zuvor.
import chainer
import chainer.links as L
import chainer.functions as F
class MLP(chainer.Chain):
def __init__(self, n_mid_units=100, n_out=10):
super(MLP, self).__init__(
l1=L.Linear(None, n_mid_units),
l2=L.Linear(n_mid_units, n_mid_units),
l3=L.Linear(n_mid_units, n_out),
)
def __call__(self, x):
h1 = F.relu(self.l1(x))
h2 = F.relu(self.l2(h1))
return self.l3(h2)
gpu_id = 0
model = MLP()
model.to_gpu(gpu_id) #Wenn Sie eine CPU verwenden, kommentieren Sie hier.
4. Vorbereitung des Updaters
Trainer ist eine Klasse, die alles zusammenstellt, was Sie zum Lernen brauchen. Der Trainer und seine internen Dienstprogrammklassen, Modelle, Datensatzklassen usw. haben die folgende Beziehung.

Wenn Sie ein "Trainer" -Objekt erstellen, übergeben Sie im Grunde nur "Updater", aber "Updater" enthält "Iterator" und "Optimizer". Sie können über Iterator auf das Dataset zugreifen, und Optimizer enthält einen Verweis auf das darin enthaltene Modell, sodass Sie die Parameter des Modells aktualisieren können. Das heißt, "Updater" ist intern
- Extrahieren Sie Daten aus dem Datensatz (Iterator)
- Übergeben Sie es an das Modell und berechnen Sie den Verlust (Model = Optimizer.target)
- Aktualisieren Sie die Modellparameter mit dem Optimizer (Optimizer).
Dies bedeutet, dass Sie den Hauptteil der Lernreihe ausführen können. Jetzt erstellen wir ein Updater-Objekt.
from chainer import optimizers
from chainer import training
max_epoch = 10
gpu_id = 0
#Wickeln Sie das Modell in Classifier ein und fügen Sie die Verlustberechnung usw. in das Modell ein
model = L.Classifier(model)
model.to_gpu(gpu_id) #Wenn Sie die CPU verwenden, kommentieren Sie diese Zeile aus.
#Auswahl der Optimierungsmethode
optimizer = optimizers.SGD()
optimizer.setup(model)
#Übergeben Sie Iterator und Optimizer an Updater
updater = training.StandardUpdater(train_iter, optimizer, device=gpu_id)
NOTE
Jetzt übergeben wir das oben definierte Modellobjekt an "L.Classifier", um es zu einer neuen "Kette" zu machen. L.Classifier ist eine Klasse, die von Chain erbt und die übergebene Chain in einer Eigenschaft namens Predictor speichert. Wenn Sie Daten und ein Label mit dem Accessor "()" übergeben, wird "call" im Inneren ausgeführt. Zuerst werden die übergebenen Daten zusammen mit den Daten durch "Predictor", ihre Ausgabe "y" und "call" geleitet. Übergibt das an die Verlustfunktion übergebene Label an die durch das Argument "lossfun" des Konstruktors angegebene Verlustfunktion und gibt die Ausgabe "Variable" zurück. lossfun ist standardmäßig in softmax_cross_entropy angegeben.
StandardUpdater ist die einfachste Klasse, um die Verarbeitung durchzuführen, für die Updater verantwortlich ist, wie oben beschrieben. Darüber hinaus werden "Parallel Updater" usw. für die Verwendung mehrerer GPUs vorbereitet.
5. Trainereinstellungen
Stellen Sie zum Schluss den Trainer ein. Beim Erstellen eines "Trainer" -Objekts ist nur das zuvor erstellte "Updater" -Objekt erforderlich, aber das zweite Argument "stop_trigger" gibt an, wann das Training beendet werden soll "(Länge ,, Wenn Sie einen Taple in Form einer Einheit geben, können Sie das Lernen automatisch zum angegebenen Zeitpunkt beenden. Die Länge kann eine beliebige Ganzzahl sein und die Einheit kann entweder "Epoche" oder "Iteration" sein. Wenn Sie nicht "stop_trigger" angeben, wird das Lernen nicht automatisch gestoppt.
#Übergeben Sie den Updater an den Trainer
trainer = training.Trainer(updater, (max_epoch, 'epoch'),
out='mnist_result')
Im Argument "out" wird das Verzeichnis zum Speichern der Protokolldatei und der Bilddatei des Diagramms, die den Prozess der Verluständerung darstellen, mit der unten beschriebenen "Erweiterung" angegeben.
6. Fügen Sie dem Trainer eine Erweiterung hinzu
Der Vorteil der Verwendung von "Trainer" ist
- Speichern Sie Protokolle automatisch in einer Datei (
LogReport) - Zeigen Sie regelmäßig Informationen wie Verluste auf dem Terminal an (
PrintReport) - Visualisieren Sie den Verlust periodisch als Grafik und speichern Sie ihn als Bild (
PlotReport) - Automatisieren Sie das Modell und den Optimierungsstatus regelmäßig automatisch (
snapshot/snapshot_object). - Zeigen Sie einen Fortschrittsbalken an, der den Lernfortschritt anzeigt (
ProgressBar) - Modellstruktur im Graphviz-Punktformat speichern (
dump_graph)
Es gibt einen Punkt, an dem Sie verschiedene praktische Funktionen wie z. Um diese Funktionen zu nutzen, übergeben Sie einfach das "Extension" -Objekt, das Sie dem "Trainer" -Objekt hinzufügen möchten, mit der "Extend" -Methode. Fügen wir nun tatsächlich einige "Erweiterungen" hinzu.
from chainer.training import extensions
trainer.extend(extensions.LogReport())
trainer.extend(extensions.snapshot(filename='snapshot_epoch-{.updater.epoch}'))
trainer.extend(extensions.snapshot_object(model.predictor, filename='model_epoch-{.updater.epoch}'))
trainer.extend(extensions.Evaluator(test_iter, model, device=gpu_id))
trainer.extend(extensions.PrintReport(['epoch', 'main/loss', 'main/accuracy', 'validation/main/loss', 'validation/main/accuracy', 'elapsed_time']))
trainer.extend(extensions.PlotReport(['main/loss', 'validation/main/loss'], x_key='epoch', file_name='loss.png'))
trainer.extend(extensions.PlotReport(['main/accuracy', 'validation/main/accuracy'], x_key='epoch', file_name='accuracy.png'))
trainer.extend(extensions.dump_graph('main/loss'))
LogReport
Es aggregiert automatisch "Verlust", "Genauigkeit" usw. für jede "Epoche" und "Iteration" und speichert sie in dem durch das "out" -Argument von "Trainer" angegebenen Ausgabeverzeichnis mit dem Dateinamen "log".
snapshot
Das "Trainer" -Objekt wird zum angegebenen Zeitpunkt (standardmäßig alle 1 Epoche) in dem durch das "out" -Argument von "Trainer" angegebenen Ausgabeverzeichnis gespeichert. Das "Trainer" -Objekt hat einen "Updater", wie oben beschrieben, und der "Optimierer" und das Modell befinden sich darin. Wenn Sie also einen Schnappschuss mit dieser "Erweiterung" machen, können Sie zum Lernen zurückkehren oder lernen. Rückschlüsse auf ein abgeschlossenes Modell sind auch nach Abschluss des Lernens möglich.
snapshot_object
Wenn Sie jedoch den gesamten "Trainer" speichern, kann es oft mühsam sein, nur das Modell im Inneren abzurufen. Daher sollten nur die mit snapshot_object angegebenen Objekte (hier das in Classifier eingeschlossene Modell) getrennt von Trainer gespeichert werden. Classifier ist eineChain, die das an das erste Argument übergebene Chain-Objekt als eigene Eigenschaft namens Predictor enthält und den Verlust berechnet, und Classifier hat überhaupt keine anderen Parameter als das Modell. Daher wird hier "model.predictor" als Speicherziel angegeben, um das trainierte Modell später für Inferenzen verwenden zu können.
dump_graph
Speichert das Berechnungsdiagramm, das vom angegebenen Variablenobjekt im Graphviz-Punktformat verfolgt werden kann. Das Speicherziel ist das Ausgabeverzeichnis, das durch das Argument "out" von "Trainer" angegeben wird.
Evaluator
Durch Übergeben des "Iterators" des Bewertungsdatensatzes und des Objekts des für das Training verwendeten Modells wird das zu trainierende Modell unter Verwendung des Bewertungsdatensatzes zum angegebenen Zeitpunkt bewertet.
PrintReport
Gibt die von "Reporter" aggregierten Werte an die Standardausgabe aus. Zu diesem Zeitpunkt wird der auszugebende Wert in Form einer Liste angegeben.
PlotReport
Zeichnet den Übergang des in der Argumentliste im Diagramm angegebenen Werts mithilfe der Bibliothek "matplotlib" und speichert ihn als Bild im Ausgabeverzeichnis mit dem durch das Argument "Dateiname" angegebenen Dateinamen.
Zusätzlich zu den hier vorgestellten haben diese "Erweiterungen" verschiedene Optionen, z. B. die Möglichkeit, festzulegen, wann sie einzeln mit dem "Auslöser" betrieben werden sollen, um mehr Flexibilität bei der Kombination zu ermöglichen. Weitere Informationen finden Sie in der offiziellen Dokumentation: Trainer-Erweiterungen.
7. Beginnen Sie zu lernen
Um mit dem Lernen zu beginnen, rufen Sie einfach die Methode "run" des "Trainer" -Objekts auf.
trainer.run()
Ausgabeergebnis
epoch main/loss main/accuracy validation/main/loss validation/main/accuracy elapsed_time
1 1.6035 0.61194 0.797731 0.833564 2.98546
2 0.595589 0.856793 0.452023 0.88123 5.74528
3 0.4241 0.885944 0.368583 0.897943 8.34872
4 0.367762 0.897152 0.33103 0.905756 11.4449
5 0.336136 0.904967 0.309321 0.912282 14.2671
6 0.314134 0.910464 0.291451 0.914557 17.0762
7 0.297581 0.914879 0.276472 0.920985 19.8298
8 0.283512 0.918753 0.265166 0.923655 23.2033
9 0.271917 0.922125 0.254976 0.926523 26.1452
10 0.260754 0.925123 0.247672 0.927413 29.3136
Ich konnte das gleiche Ergebnis mit umfangreichen Protokollinformationen und einem Diagramm wie dem unten gezeigten erzielen, mit einem kürzeren Code, als wenn ich die Lernschleife geschrieben hätte, an der ich zu Beginn gearbeitet habe.
Lassen Sie uns das Diagramm für gespeicherte Verluste sofort überprüfen.
from IPython.display import Image
Image(filename='mnist_result/loss.png')
\ * Dieser Teil muss auf dem Jupyter-Notebook ausgeführt werden, um die folgenden Ergebnisse zu erhalten.
Ausgabeergebnis

Schauen wir uns auch das Genauigkeitsdiagramm an.
Image(filename='mnist_result/accuracy.png')
Ausgabeergebnis
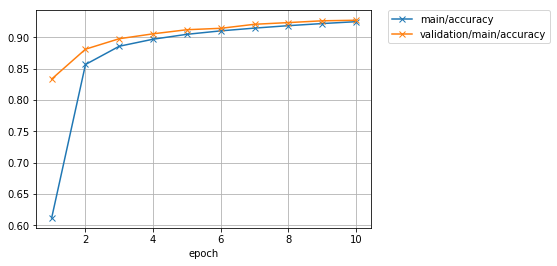
Wenn Sie weiter lernen, gibt es immer noch eine Atmosphäre, in der Sie die Genauigkeit ein wenig verbessern können.
Als nächstes verwenden wir Graphviz, um das von Extension ausgegebene Berechnungsdiagramm zu visualisieren, das als dump_graph bezeichnet wird.
%%bash
dot -Tpng mnist_result/cg.dot -o mnist_result/cg.png
\ * Hier verwenden wir Cell Magic, das den Befehl bash auf dem Jupyter-Notizbuch verwendet. Der Befehl in der zweiten Zeile selbst ist ein normaler Shell-Befehl.
Image(filename='mnist_result/cg.png')
Ausgabeergebnis

Von oben nach unten können Sie sehen, an welche Funktion die Daten und Parameter für die Berechnung übergeben wurden und welche Variable für den Verlust ausgegeben wurde.
8. Schliessen Sie mit einem trainierten Modell
import numpy as np
from chainer import serializers
from chainer.cuda import to_gpu
from chainer.cuda import to_cpu
model = MLP()
serializers.load_npz('mnist_result/model_epoch-10', model)
model.to_gpu(gpu_id)
%matplotlib inline
import matplotlib.pyplot as plt
x, t = test[0]
plt.imshow(x.reshape(28, 28), cmap='gray')
plt.show()
print('label:', t)
x = to_gpu(x[None, ...])
y = model(x)
y = to_cpu(y.data)
print('predicted_label:', y.argmax(axis=1)[0])
Ausgabeergebnis

label: 7
predicted_label: 7
Ich konnte richtig antworten.
Schreiben wir ein neues Netzwerk
Versuchen Sie hier, anstatt das MNIST-Dataset zu verwenden, verschiedene Modelle selbst mit einem kleinen Farbbild der Größe 32 x 32 mit der Bezeichnung CIFAR10 zu schreiben, das mit einer der 10 Klassen gekennzeichnet ist, und erleben Sie den Ablauf von Versuch und Irrtum. Ich werde.
| airplane | automobile | bird | cat | deer | dog | frog | horse | ship | truck |
|---|---|---|---|---|---|---|---|---|---|
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
1. Modelldefinition
Das Modell wird durch Erben der Klasse "Chain" definiert. Definieren wir hier ein Netzwerk mit einer Faltungsschicht anstelle des Netzwerks, das nur aus den vollständig verbundenen Schichten besteht, die wir zuvor versucht haben. Dieses Modell hat drei Faltungsschichten, gefolgt von zwei vollständig verbundenen Schichten.
Das Modell wird hauptsächlich durch die Definition von zwei Methoden definiert.
- Definieren Sie die Ebenen, aus denen das Modell besteht, mit dem Konstruktor
__init__
- Zu diesem Zeitpunkt eine Ebene mit Optimierungszielparametern, die von "Optimizer" erfasst werden können, indem ein "Link" -Objekt, das das Modell als Schlüsselwortargument mit "super" zusammensetzt, an den Konstruktor der übergeordneten Klasse ("Chain") übergeben wird. Kann dem Modell hinzugefügt werden.
- Schreiben Sie die Vorwärtsberechnung in die Methode
__call__, die vom()Accessor aufgerufen wird, der die Daten empfängt.
import chainer
import chainer.functions as F
import chainer.links as L
class MyModel(chainer.Chain):
def __init__(self, n_out):
super(MyModel, self).__init__(
conv1=L.Convolution2D(None, 32, 3, 3, 1),
conv2=L.Convolution2D(32, 64, 3, 3, 1),
conv3=L.Convolution2D(64, 128, 3, 3, 1),
fc4=L.Linear(None, 1000),
fc5=L.Linear(1000, n_out)
)
def __call__(self, x):
h = F.relu(self.conv1(x))
h = F.relu(self.conv2(h))
h = F.relu(self.conv3(h))
h = F.relu(self.fc4(h))
h = self.fc5(h)
return h
2. Lernen
Definieren Sie hier die Funktion "Zug", damit Sie später problemlos ein anderes Modell mit denselben Einstellungen trainieren können. das ist,
- Modellobjekt
- Stapelgröße
- GPU ID zu verwenden
- Anzahl der Epochen, um das Lernen zu beenden --Dataset-Objekt
Wenn Sie bestehen, ist es eine Funktion, die das Modell intern anhand des mit "Trainer" übergebenen Datensatzes trainiert und das Modell in dem Zustand zurückgibt, in dem das Training abgeschlossen ist.
Verwenden wir diese "Zug" -Funktion, um das oben definierte "MyModel" -Modell zu trainieren.
from chainer.datasets import cifar
from chainer import iterators
from chainer import optimizers
from chainer import training
from chainer.training import extensions
def train(model_object, batchsize=64, gpu_id=0, max_epoch=20, train_dataset=None, test_dataset=None):
# 1. Dataset
if train_dataset is None and test_dataset is None:
train, test = cifar.get_cifar10()
else:
train, test = train_dataset, test_dataset
# 2. Iterator
train_iter = iterators.SerialIterator(train, batchsize)
test_iter = iterators.SerialIterator(test, batchsize, False, False)
# 3. Model
model = L.Classifier(model_object)
if gpu_id >= 0:
model.to_gpu(gpu_id)
# 4. Optimizer
optimizer = optimizers.Adam()
optimizer.setup(model)
# 5. Updater
updater = training.StandardUpdater(train_iter, optimizer, device=gpu_id)
# 6. Trainer
trainer = training.Trainer(updater, (max_epoch, 'epoch'), out='{}_cifar10_result'.format(model_object.__class__.__name__))
# 7. Evaluator
class TestModeEvaluator(extensions.Evaluator):
def evaluate(self):
model = self.get_target('main')
model.train = False
ret = super(TestModeEvaluator, self).evaluate()
model.train = True
return ret
trainer.extend(extensions.LogReport())
trainer.extend(TestModeEvaluator(test_iter, model, device=gpu_id))
trainer.extend(extensions.PrintReport(['epoch', 'main/loss', 'main/accuracy', 'validation/main/loss', 'validation/main/accuracy', 'elapsed_time']))
trainer.extend(extensions.PlotReport(['main/loss', 'validation/main/loss'], x_key='epoch', file_name='loss.png'))
trainer.extend(extensions.PlotReport(['main/accuracy', 'validation/main/accuracy'], x_key='epoch', file_name='accuracy.png'))
trainer.run()
del trainer
return model
model = train(MyModel(10), gpu_id=0) #Beim Laufen auf der CPU`gpu_id=-1`Bitte angeben.
Ausgabeergebnis
epoch main/loss main/accuracy validation/main/loss validation/main/accuracy elapsed_time
1 1.53309 0.444293 1.29774 0.52707 5.2449
2 1.21681 0.56264 1.18395 0.573746 10.6833
3 1.06828 0.617358 1.10173 0.609773 16.0644
4 0.941792 0.662132 1.0695 0.622611 21.2535
5 0.832165 0.703345 1.0665 0.624104 26.4523
6 0.729036 0.740257 1.0577 0.64371 31.6299
7 0.630143 0.774208 1.07577 0.63953 36.798
8 0.520787 0.815541 1.15054 0.639431 42.1951
9 0.429535 0.849085 1.23832 0.6459 47.3631
10 0.334665 0.882842 1.3528 0.633061 52.5524
11 0.266092 0.90549 1.44239 0.635251 57.7396
12 0.198057 0.932638 1.6249 0.6249 62.9918
13 0.161151 0.944613 1.76964 0.637241 68.2177
14 0.138705 0.952145 1.98031 0.619725 73.4226
15 0.122419 0.957807 2.03002 0.623806 78.6411
16 0.109989 0.962148 2.08948 0.62281 84.3362
17 0.105851 0.963675 2.31344 0.617237 89.5656
18 0.0984753 0.966289 2.39499 0.624801 95.1304
19 0.0836834 0.970971 2.38215 0.626791 100.36
20 0.0913404 0.96925 2.46774 0.61873 105.684
Das Lernen dauert bis zu 20 Epochen. Werfen wir einen Blick auf das Verlust- und Genauigkeitsdiagramm.
Image(filename='MyModel_cifar10_result/loss.png')

Image(filename='MyModel_cifar10_result/accuracy.png')

Die Genauigkeit ("Haupt / Genauigkeit") in den Trainingsdaten hat ungefähr 97% erreicht, aber der Verlust ("Validierung / Haupt / Verlust") in den Testdaten hat mit jedem Iterationsfortschritt eher zugenommen. Auch die Genauigkeit der Testdaten ("Validierung / Haupt / Genauigkeit") hat einen Höchstwert von rund 62% erreicht. Es scheint, dass das ** Modell die Trainingsdaten ** überpasst, da die Trainingsdaten eine gute Genauigkeit aufweisen, die Testdaten jedoch nicht.
3. Vorhersage unter Verwendung eines trainierten Modells
Die Testgenauigkeit lag bei 62%. Versuchen wir jedoch, mit diesem trainierten Modell einige Testbilder zu klassifizieren.
%matplotlib inline
import matplotlib.pyplot as plt
cls_names = ['airplane', 'automobile', 'bird', 'cat', 'deer',
'dog', 'frog', 'horse', 'ship', 'truck']
def predict(model, image_id):
_, test = cifar.get_cifar10()
x, t = test[image_id]
model.to_cpu()
y = model.predictor(x[None, ...]).data.argmax(axis=1)[0]
print('predicted_label:', cls_names[y])
print('answer:', cls_names[t])
plt.imshow(x.transpose(1, 2, 0))
plt.show()
for i in range(10, 15):
predict(model, i)
Ausgabeergebnis
predicted_label: dog
answer: airplane

predicted_label: truck
answer: truck

predicted_label: bird
answer: dog

predicted_label: horse
answer: horse

predicted_label: truck
answer: truck

Einige waren gut kategorisiert, andere nicht. Selbst wenn die richtige Antwort in fast 100 Aufnahmen des für das Training des Modells verwendeten Datensatzes erhalten werden kann, ist dies bedeutungslos, es sei denn, die unbekannten Daten, dh das Bild im Testdatensatz, können mit hoher Genauigkeit vorhergesagt werden [^ NN]. Die Genauigkeit der Testdaten soll mit der ** Generalisierungsleistung ** des Modells zusammenhängen.
Wie können Sie ein Modell mit hoher Generalisierungsleistung entwerfen und trainieren?
4. Definieren wir ein tieferes Modell
Definieren wir nun ein Modell mit mehr Ebenen als das obige Modell. Hier definieren wir ein einschichtiges gewundenes Netzwerk als "ConvBlock" und ein einschichtiges, vollständig verbundenes Netzwerk als "LinearBlock" und definieren ein großes Netzwerk, indem wir viele davon nacheinander stapeln.
Komponenten definieren
Definieren wir zunächst "ConvBlock" und "LinearBlock", die die Komponenten des großen Netzwerks sind, das wir anstreben.
class ConvBlock(chainer.Chain):
def __init__(self, n_ch, pool_drop=False):
w = chainer.initializers.HeNormal()
super(ConvBlock, self).__init__(
conv=L.Convolution2D(None, n_ch, 3, 1, 1,
nobias=True, initialW=w),
bn=L.BatchNormalization(n_ch)
)
self.train = True
self.pool_drop = pool_drop
def __call__(self, x):
h = F.relu(self.bn(self.conv(x)))
if self.pool_drop:
h = F.max_pooling_2d(h, 2, 2)
h = F.dropout(h, ratio=0.25, train=self.train)
return h
class LinearBlock(chainer.Chain):
def __init__(self):
w = chainer.initializers.HeNormal()
super(LinearBlock, self).__init__(
fc=L.Linear(None, 1024, initialW=w))
self.train = True
def __call__(self, x):
return F.dropout(F.relu(self.fc(x)), ratio=0.5, train=self.train)
"ConvBlock" ist ein Modell, das "Chain" erbt. Es hat eine Faltungsschicht und eine Chargennormalisierungsschicht mit Parametern, daher registrieren wir diese im Konstruktor. Bei der Methode __call__ wird beim Übergeben von Daten an diese die Aktivierungsfunktion ReLU angewendet, und wenn pool_drop mit True an den Konstruktor übergeben wird, werden die Funktionen Max Pooling und Dropout angewendet. Es ist ein kleines Netzwerk.
In Chainer repräsentiert der mit Python selbst geschriebene Vorwärtsberechnungscode die Struktur des Modells. Mit anderen Worten, welche Schicht die Daten zur Laufzeit durchlaufen haben, definiert das Netzwerk selbst. Dies erleichtert das Schreiben von Netzwerken mit Verzweigungen wie oben beschrieben und ermöglicht flexible, einfache und gut lesbare Netzwerkdefinitionen. Dies ist eine Funktion namens ** Define-by-Run **.
Definition eines großen Netzwerks
Als nächstes stapeln wir diese kleinen Netzwerke als Komponenten, um ein großes Netzwerk zu definieren.
class DeepCNN(chainer.ChainList):
def __init__(self, n_output):
super(DeepCNN, self).__init__(
ConvBlock(64),
ConvBlock(64, True),
ConvBlock(128),
ConvBlock(128, True),
ConvBlock(256),
ConvBlock(256),
ConvBlock(256),
ConvBlock(256, True),
LinearBlock(),
LinearBlock(),
L.Linear(None, n_output)
)
self._train = True
@property
def train(self):
return self._train
@train.setter
def train(self, val):
self._train = val
for c in self.children():
c.train = val
def __call__(self, x):
for f in self.children():
x = f(x)
return x
Die Klasse, die wir hier verwenden, ist "ChainList". Diese Klasse erbt von "Chain" und ist nützlich, wenn Sie ein Netzwerk definieren, das mehrere "Links" und "Chain" nacheinander aufruft. Modelle, die durch das Erben von "ChainList" definiert wurden, können beim Aufruf des Konstruktors der übergeordneten Klasse ** "Link" - oder "Chain" -Objekte als reguläre Argumente anstelle von ** Schlüsselwortargumenten übergeben. Und diese können in der Reihenfolge, in der sie registriert wurden, mit der Methode self.children () abgerufen werden.
Diese Funktion erleichtert das Schreiben von Vorwärtsberechnungen. Aus der Liste der von ** self.children () ** zurückgegebenen Komponenten werden die Komponenten in der Reihenfolge durch die for-Anweisung extrahiert, und die extrahierte partielle Netzwerkberechnung wird auf die ursprüngliche Eingabe "x" angewendet. Wenn Sie "x" in der angegebenen Reihenfolge durch diese Ausgabe ersetzen, können Sie eine Reihe von "Link" oder "Chain" in derselben Reihenfolge anwenden, wie sie in der übergeordneten Klasse im Konstruktor registriert ist. Daher ist es nützlich, ein großes Netzwerk zu definieren, das durch die Anwendung von sequentiellen Teilnetzwerken dargestellt wird.
Jetzt lasst uns lernen. Da es diesmal viele Parameter gibt, stellen Sie die Anzahl der Epochen, in denen das Lernen beendet werden soll, auf 100 ein.
model = train(DeepCNN(10), max_epoch=100)
Ausgabeergebnis
epoch main/loss main/accuracy validation/main/loss validation/main/accuracy elapsed_time
1 2.05147 0.242887 1.71868 0.340764 14.8099
2 1.5242 0.423816 1.398 0.48537 29.12
3 1.24906 0.549096 1.12884 0.6042 43.4423
4 0.998223 0.652649 0.937086 0.688495 58.291
5 0.833486 0.720009 0.796678 0.73756 73.4144
.
.
.
95 0.0454193 0.987616 0.815549 0.863555 1411.86
96 0.0376641 0.990057 0.878458 0.873109 1426.85
97 0.0403836 0.98953 0.849209 0.86465 1441.19
98 0.0369386 0.989677 0.919462 0.873905 1456.04
99 0.0361681 0.990677 0.88796 0.86873 1470.46
100 0.0383634 0.988676 0.92344 0.869128 1484.91
(Da das Protokoll lang ist, wird die Mitte weggelassen.)
Das Lernen ist beendet. Werfen wir einen Blick auf die Verlust- und Genauigkeitsdiagramme.
Image(filename='DeepCNN_cifar10_result/loss.png')

Image(filename='DeepCNN_cifar10_result/accuracy.png')

Sie sehen, dass sich die Genauigkeit der Testdaten im Vergleich zum vorherigen Zeitpunkt erheblich verbessert hat. Die Genauigkeit von rund 62% hat sich auf rund 87% erhöht. Die neuesten Forschungsergebnisse haben jedoch fast 97% erreicht. Um die Genauigkeit weiter zu verbessern, wird nicht nur das Modell dieses Mal verbessert, sondern auch die Trainingsdaten künstlich erhöht (Datenerweiterung) und die Ausgaben mehrerer Modelle in eine Ausgabe integriert (Ensemble). Und so weiter können verschiedene Ideen berücksichtigt werden.
Schreiben wir eine Datensatzklasse
Hier schreibe ich die Datensatzklasse selbst mit der Funktion, um die Daten von CIFAR10 zu erfassen, die bereits in Chainer vorbereitet wurden. Für Chainer muss die Klasse, die das Dataset darstellt, über die folgenden Funktionen verfügen:
--__len__ Methode, die die Anzahl der Daten im Dataset zurückgibt
- Die Methode "get_example", die die Daten oder das Daten / Label-Paar zurückgibt, die dem als Argument übergebenen "i" entsprechen.
Die für andere Datasets erforderliche Funktionalität kann durch Erben der Klasse "chainer.dataset.DatasetMixin" bereitgestellt werden. Hier erstellen wir eine Dataset-Klasse mit der Datenerweiterungsfunktion, indem wir die DatasetMixin-Klasse erben.
1. Schreiben Sie eine CIFAR10-Dataset-Klasse
import numpy as np
from chainer import dataset
from chainer.datasets import cifar
class CIFAR10Augmented(dataset.DatasetMixin):
def __init__(self, train=True):
train_data, test_data = cifar.get_cifar10()
if train:
self.data = train_data
else:
self.data = test_data
self.train = train
self.random_crop = 4
def __len__(self):
return len(self.data)
def get_example(self, i):
x, t = self.data[i]
if self.train:
x = x.transpose(1, 2, 0)
h, w, _ = x.shape
x_offset = np.random.randint(self.random_crop)
y_offset = np.random.randint(self.random_crop)
x = x[y_offset:y_offset + h - self.random_crop,
x_offset:x_offset + w - self.random_crop]
if np.random.rand() > 0.5:
x = np.fliplr(x)
x = x.transpose(2, 0, 1)
return x, t
Diese Klasse gilt für alle CIFAR10-Daten
- 28x28 Fläche zufällig aus 32x32 Größe zuschneiden
- Invertieren Sie links und rechts mit einer halben Chance
Wir machen die Verarbeitung. Es ist bekannt, dass das Erhöhen der Variation von Trainingsdaten auf Pseudo-Weise durch Hinzufügen solcher Operationen nützlich ist, um eine Überanpassung zu unterdrücken. Zusätzlich zu diesen Operationen wurden Verfahren vorgeschlagen, um die Anzahl von Trainingsdaten auf Pseudo-Weise durch verschiedene Verarbeitungen zu erhöhen, wie z. B. Konvertierung, die die Farbe des Bildes ändert, zufällige Drehung und affine Konvertierung.
Wenn Sie den Datenerfassungsteil selbst schreiben möchten, übergeben Sie den Pfad des Bildordners und den Pfad zur Textdatei mit der Bezeichnung, die dem Dateinamen entspricht, an den Konstruktor und behalten Sie ihn als Eigenschaft bei, und zwar in der Methode "get_example" Sie können sehen, dass jedes Bild gelesen und mit dem entsprechenden Etikett zurückgegeben werden sollte.
2. Lernen Sie die Verwendung der erstellten Dataset-Klasse
Beginnen wir jetzt mit dem Lernen mit dieser CIFAR10-Klasse. Lassen Sie uns herausfinden, wie effektiv die Datenerweiterung ist, indem Sie dasselbe große Netzwerk verwenden, das wir zuvor verwendet haben. Mit Ausnahme der Datensatzklasse, einschließlich der Funktion "Zug", entspricht sie fast dem zuvor verwendeten Code. Der einzige Unterschied ist die Anzahl der Epochen und der Name des Zielverzeichnisses.
model = train(DeepCNN(10), max_epoch=100, train_dataset=CIFAR10Augmented(), test_dataset=CIFAR10Augmented(False))
Ausgabeergebnis
epoch main/loss main/accuracy validation/main/loss validation/main/accuracy elapsed_time
1 2.023 0.248981 1.75221 0.322353 18.4387
2 1.51639 0.43716 1.36708 0.512639 36.482
3 1.25354 0.554177 1.17713 0.586087 54.6892
4 1.05922 0.637804 0.971438 0.665904 72.9602
5 0.895339 0.701886 0.918005 0.706409 91.4061
.
.
.
95 0.0877855 0.973171 0.726305 0.89162 1757.87
96 0.0780378 0.976012 0.943201 0.890725 1776.41
97 0.086231 0.973765 0.57783 0.890227 1794.99
98 0.0869593 0.973512 1.65576 0.878981 1813.52
99 0.0870466 0.972931 0.718033 0.891421 1831.99
100 0.079011 0.975332 0.754114 0.892815 1850.46
(Da das Protokoll lang ist, wird die Mitte weggelassen.)
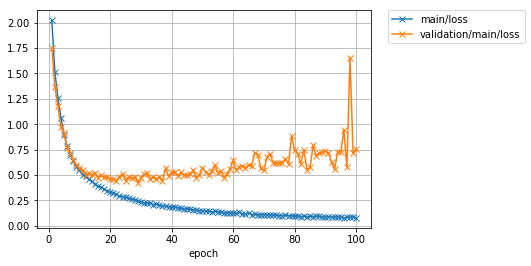
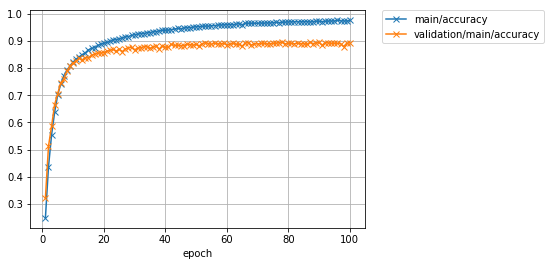
Es wurde festgestellt, dass die Genauigkeit, die ohne Datenerweiterung einen Spitzenwert von etwa 87% erreicht hatte, durch Anwenden einer Erweiterung der Trainingsdaten auf 89% oder mehr verbessert werden kann. Dies ist eine Verbesserung von etwas mehr als 2%.
Schauen wir uns zum Schluss die Verlust- und Genauigkeitsdiagramme an.
Image(filename='DeepCNN_cifar10augmented_result/loss.png')
Ausgabeergebnis
Image(filename='DeepCNN_cifar10augmented_result/accuracy.png')
abschließend
In diesem Artikel über Chainer
- Wie schreibe ich eine Lernschleife ohne Trainer
- Wie man Trainer benutzt
- So schreiben Sie Ihr eigenes Modell
- So schreiben Sie Ihre eigene Datensatzklasse
Wurde kurz vorgestellt. Ich weiß nicht, ob es in Zukunft im Hands-on-Format gemacht wird, aber ich möchte einen Kommentar zu so etwas wie dem Folgenden schreiben.
--Wie erstelle ich meinen eigenen Updater und Iterator, aus denen der Trainer besteht?
- So optimieren Sie vorab trainierte Modelle wie "VGG16Layers" und "ResNet50Layers" unter dem Modul "chainer.links.models.vision" für bestimmte Aufgaben
- So machen Sie eine Erweiterung
Wir begrüßen auch alle, die sich für die Entwicklung von Chainer engagieren! Chainer ist Open-Source-Software, daher werden wir uns weiterentwickeln, indem wir die gewünschten Funktionen vorschlagen und Pull-Anfragen senden. Wenn Sie interessiert sind, lesen Sie bitte diesen Contoribution Guide und machen Sie dann ein Problem oder senden Sie eine PR. schauen Sie bitte. Wir werden dich erwarten.
pfent/chainer https://github.com/pfnet/chainer
Fußnote
[^ Trainings- und Validierungsdaten]: Dieser Artikel konzentriert sich auf die Erläuterung der Verwendung von Chainer, sodass nicht klar zwischen den Validierungs- und Testdatensätzen unterschieden wird. In Wirklichkeit sollten diese jedoch unterschieden werden. Normalerweise entfernen Sie einige der Trainingsdaten aus dem Trainingsdatensatz und konfigurieren den Validierungsdatensatz mit den entfernten Daten. Danach besteht das allgemeine Verfahren darin, zuerst das mit den Trainingsdaten trainierte Modell mit den Validierungsdaten auszuwerten und dann das Modell zu verbessern, um die Leistung mit den Validierungsdaten zu verbessern. Testdaten werden erst verwendet, um die Leistung des endgültigen Modells zu bewerten (z. B. zum Vergleich mit anderen Modellen), nachdem alle Bemühungen abgeschlossen wurden. In einigen Fällen können mehrere Trainings- / Validierungsdatenkonfigurationen vorbereitet werden, um eine Überanpassung aufgrund einer Modellverbesserung unter Verwendung verzerrter Daten zu vermeiden. [^ NN]: Die Vorhersagegenauigkeit für Trainingsdaten sind Daten, die gefunden werden können, indem bestimmte aus Trainingsdaten extrahierte Daten abgefragt und aus dem Trainingsdatensatz gesucht werden, der sie enthält. Wenn Sie das beigefügte Etikett beantworten, erhalten Sie 100%.
Recommended Posts