[PYTHON] National Medical AI Contest 2020 Lösung für den 1. Platz
0. Einleitung
Ich habe den nationalen medizinischen KI-Wettbewerb 2020 gewonnen, einen von der AI & Machine Learning Society (AIMS) der Universität Osaka gesponserten Wettbewerb für medizinische Tischdaten (1. Platz, Top 6%), daher werde ich die Lösung veröffentlichen.
Wie Sie vielleicht aus den bisherigen Artikeln wissen, bin ich im Tischdatenwettbewerb nicht sehr gut, weil ich mich auf Bilddaten spezialisiert habe. Bitte beachten Sie, dass es einige Fehler und irrationale Teile geben kann.
Darüber hinaus richtet sich dieser Artikel grundsätzlich an diejenigen, die am Wettbewerb teilgenommen haben. Es gibt keine Erklärung zu bestimmten Spaltennamen und Feature-Mengen, sodass Sie diese Teile überspringen können. (Der technische Teil wird ausführlich erklärt!)
Da es sich um einen privaten Wettbewerb handelt, können Sie nur auf die Teilnehmer des Wettbewerbs zugreifen.
1. Über den Wettbewerb
1-1. Überblick über den Wettbewerb
Der Wettbewerb ist ein Tabellendatenwettbewerb zur Vorhersage der Mortalität von COVID-19-Patienten. Wir werden Daten zu den Risikofaktoren (Ort, Vorhandensein oder Nichtvorhandensein einer Lungenentzündung, Alter usw.) analysieren, die zu einem bestimmten Tod führen können, und ein Modell erstellen und einreichen. Die Wettbewerbsdauer ist sehr kurz, 2 Tage (zwischen 9/26 14:00 und 9/27 12:00), und der Schlüssel ist, wie schnell Inferenz, Implementierung und Optimierung durchgeführt werden können. .. Darüber hinaus war die Teilnahme am Wettbewerb auf Studenten beschränkt.
1-2. Bewertungsindex
Der Bewertungsindex ist die Fläche unter der ROC-Kurve. Dieser Bewertungsindex wurde bereits im folgenden Artikel erläutert, daher werde ich ihn weglassen. [Melanom-Wettbewerbsbereich unter ROC-Kurve](https://qiita.com/tachyon777/items/05e7d35b7e0b53ef03dd#1-2%E8%A9%95%E4%BE%A1%E6%8C%87%E6%A8% 99)
1-3. Datensatz
Datensatz ist einfach ・ Train.csv ・ Test.csv ・ Sample_submission.csv Es besteht aus drei Teilen. Lernen Sie mit dem Zug, sagen Sie den Test voraus und reichen Sie ihn in Form von sample_submission ein.
In Bezug auf den Dateninhalt gab es Folgendes. ・ Patientenadresse (Details) ·Alter ・ Lage des Krankenhauses ・ Geburtsort des Patienten ・ Einrichtungen, in denen Patienten behandelt wurden ・ Haben Sie in der Vergangenheit Kontakt zu anderen COVID-Patienten? ・ PCR-Testergebnisse ·Lungenentzündung · Datum des Beginns ・ Krankenhausaufenthalt (Konsultationstermin) ・ Intubation ・ Krankenhausaufenthalt oder ambulant ・ Vorhandensein oder Nichtvorhandensein von chronischem Nierenversagen, Diabetes, Bluthochdruck, Herz-Kreislauf-Erkrankungen, Asthma und anderen Grunderkrankungen
Da sich das Ergebnis der Vorhersage von test.csv tatsächlich in LeaderBoard (nicht in Code Competition) widerspiegelt, ist die als Testdaten angegebene Menge groß, und es wurde angenommen, dass Methoden wie Pseudo-Labeling effektiv sind.
2.EDA Ich musste nichts tun. Das liegt daran, dass Akiyama, der den Wettbewerb leitet, zu Beginn des Wettbewerbs ein Notizbuch herausgebracht hat, das EDA und Baseline kombiniert, und fast alle erforderlichen Informationen verfügbar waren. Insbesondere wurde die Punktzahl von LightGBMs feature_importance aufgelistet, und der Rest diente lediglich der Implementierung des Modells. Mit einer sehr leicht verständlichen Datenanalyse konnte ich sofort mit der Implementierung des Modells beginnen.
3. Theorie
Theoretisch werde ich diesmal die Schlüsselmethode meiner Lösung erläutern.
3-1. Target Encoding Die Zielcodierung ist eine Methode zur Verwendung des "Durchschnittswerts der korrekten Beschriftungen der Kategorie" selbst als Merkmalsmenge in einer Kategorievariablen. Da das richtige Antwortetikett selbst indirekt als Merkmalsmenge verwendet wird, ist es erforderlich, vorsichtig mit Lecks umzugehen, und die Implementierung ist recht mühsam. Der Grund, warum ich diese Technik dieses Mal brauchte, war, dass es eine kategoriale Variable namens "place_patient_live2" mit 477 Kategorien gab. Wenn eine kategoriale Variable in NN verwendet wird, wird sie normalerweise von OneHotEncoding verarbeitet (in GBDT kann die kategoriale Variable unverändert verwendet werden). Wenn Sie jedoch 477 Spalten erstellen und nur eine davon sinnvoll ist, sind viele davon sinnvoll Sie können sehen, dass die Spalte zu einer nutzlosen Merkmalsmenge wird (= spärliche Matrix). Daher war es notwendig, die Anzahl der Spalten (477 → 1) durch Verwendung der Zielcodierung signifikant zu reduzieren, damit das Lernen effizient durchgeführt werden konnte. Letzteres war in meinem Modell beim Trainieren mit OneHotEncoding und beim Trainieren mit Zielcodierung wesentlich genauer.
3-2. Pseudo Labeling Pseudo-Labeling ist eine halbüberwachte Lernmethode. Wir möchten die Genauigkeit verbessern, indem wir nicht nur Trainingsdaten, sondern auch Testdaten lernen. Trainieren Sie zuerst die Trainingsdaten mit einem normalen Modell, werten Sie die Testdaten aus und geben Sie den vorhergesagten Wert aus. Verwenden Sie diesen vorhergesagten Wert jedoch als Bezeichnung für die Testdaten und trainieren Sie erneut mit Training + Testdaten von 0. Ich werde. Infolgedessen kann die Diversität der Testdaten erhalten werden, wenn das Trainingsvorhersageergebnis bis zu einem gewissen Grad korrekt ist. Wenn Sie diese Methode verwenden, müssen Sie über ausreichende Testdaten verfügen. Sie kann nicht für Wettbewerbe wie den Code-Wettbewerb verwendet werden, bei denen fast keine Testdaten angegeben werden. Sie müssen auch vorsichtig mit der Menge der von Ihnen verwendeten Testdaten sein, und es wird gesagt, dass es besser ist, eine Menge zu verwenden, bei der train: test = 2: 1.
Mit dieser Methode kann die Genauigkeit eines einzelnen Modells bestimmt werden.
- Public LB 0.96696→0.96733 Und erzielte eine signifikante Verbesserung der Genauigkeit.
Dieses Mal haben wir die Ausgabeergebnisse des NN-Modells und des LGBM-Modellensembles tatsächlich als Testdatenetiketten verwendet.
4. Überprüfung
4-1. Richtlinien
Ursprünglich hatte ich vor Beginn dieses Wettbewerbs eine ungefähre Vorstellung davon, dass es sich um Tabellendaten handeln würde. Lassen Sie uns also mit dem Ablauf von EDA fortfahren → Baseline mit LightGBM erstellen → Gewichtsextraktion der Feature-Menge → NN-Implementierung! Zu Beginn des Wettbewerbs wurden jedoch die Implementierung von EDA und LightGBM durch Herrn Akiyama, die Operation und das Gewicht der Feature-Menge veröffentlicht, sodass nicht mehr viel zu tun war. (Ich freute mich darauf, es von Grund auf neu zu implementieren, daher ist es ein bisschen enttäuschend für diejenigen, die Konkurrenz erlebt haben ...)
Nachdem wir die Genauigkeit von LightGBM kennen, haben wir Pytorch verwendet, um das lineare Modell als Basis zu implementieren. (Vorerst bestand die Richtlinie darin, Feature-Mengen mit LightGBM so wie sie sind zu erstellen. Da ich jedoch nicht viel über Feature-Engineering weiß, habe ich plötzlich mit der Erstellung von NN begonnen.)
4-2. Auswahl und Verwendung der zu verwendenden Funktionen
Für die zu verwendende Funktionsmenge habe ich fast alle bereits veröffentlichten LGBM-Modelle verwendet. Da die Methode zum Erstellen des anzuwendenden Merkmalsbetrags für jeden unterschiedlich ist, wird der an der Basislinie verwendete im Folgenden grob beschrieben.
** ・ Standardisierung ** Standardisierung. Skalieren Sie auf Durchschnitt 0, Varianz 1.
standard_cols = [
"age",
"entry_-_symptom_date",
"entry_date_count",
"date_symptoms",
"entry_date",
]
** ・ Onehot-Codierung ** Verarbeitung kategorialer Variablen. Erstellen Sie so viele Spalten wie es Kategorien gibt, und setzen Sie 1 für diese Spalte und 0 für andere.
onehot_cols = [
"place_hospital",
"place_patient_birth",
"type_hospital",
"contact_other_covid",
"test_result",
"pneumonia",
"intubed",
"patient_type",
"chronic_renal_failure",
"diabetes",
"icu",
"obesity",
"immunosuppression",
"sex",
"other_disease",
"pregnancy",
"hypertension",
"cardiovascular",
"asthma",
"tobacco",
"copd",
]
** ・ Zielcodierung ** Eine Methode, die den Durchschnitt der korrekten Beschriftungen für die gesamte Kategorie als Feature-Menge verwendet target_E_cols = ["place_patient_live2"]
・ Datumsdaten Ändern Sie zunächst die als Datum (26.09.2018) angegebenen Daten in Daten, die als kontinuierlicher Wert betrachtet werden können (Beispiel: 9/27: 1, 9/28: 2, wenn 9/26 0 ist). .. Danach wird min_max_encoding ausgeführt, wobei der Maximalwert auf 1 und der Minimalwert auf 0 gesetzt wird.
4-3. Grundlinie
name : medcon2020_tachyon_baseline
about : simple NN baseline
model : Liner Model
batch : 32
epoch : 20
criterion : BCEWithLogitsLoss
optimizer : Adam
init_lr : 1e-2
scheduler: CosineAnnealingLR
data : plane
preprocess : OnehotEncoding,Standardization,Target Encoding
train_test_split : StratifiedKFold, k=5
Public LB : 0.96575
4-4. LightGBM-Modell
Was die Merkmalsmenge betrifft, wurde die Parameteroptimierung durchgeführt, indem Optuna unter Verwendung des Merkmalsentwicklungsergebnisses von Herrn Akiyama angewendet wurde. Ergebnis,
Akiyamas Grundlinie: Public LB0.96636
Modell mit Optuna: Public LB0.96635
Da fast das gleiche Ergebnis erzielt wurde, kann gesagt werden, dass diese Basislinie ein ziemlich vollständiges Modell war. Da die Modelle jedoch teilweise unterschiedliche Parameter aufweisen, ist der Ensemble-Effekt (Verbesserung der Genauigkeit aufgrund von Diversität) in gewissem Maße zu erwarten.
4-5. Einreichung des Ensembles
Nachdem ich die Basislinienparameter von 4-2 angepasst und eine gewisse Genauigkeit erhalten hatte, reichte ich zuerst ein NN-Modell und Akiyamas durchschnittliches Ensemble für das Basislinienmodell ein.
※Public LB
NN allein: 0.96696
LGBM allein: 0.96636
Average Ensembling : 0.96738
Es ist eine unerwartete Verbesserung der Genauigkeit. Da die Algorithmen von NN und GBDT völlig unterschiedlich sind, wird angenommen, dass die Punktzahl signifikant gestiegen ist. Zu dieser Zeit war ich die Nummer eins in Butchigiri.
4-6.Pseudo Labeling Am zweiten Tag (letzter Tag) haben wir die in 3. Theorie erläuterte Pseudo-Kennzeichnung implementiert. Es gab vielleicht einen schnelleren Weg, um die Genauigkeit zu verbessern, aber dies war der einzige, der mir in den Sinn kam ...
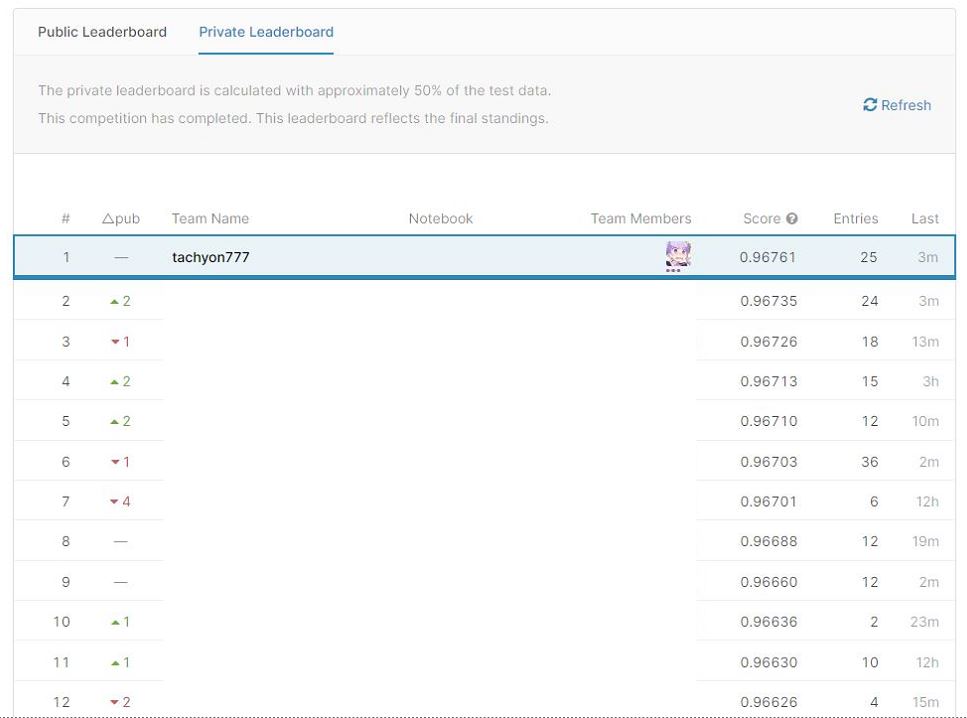
Vor der Implementierung der Pseudo-Kennzeichnung Durchschnittlicher Ensembling-Score: Privat: 0,96753 Öffentlich: 0,96761 Nach der Implementierung von Pseudo Labeling Average Ensembling Score: ** Privat: 0,96761 ** Öffentlich: 0,96768
Immerhin habe ich diese beiden als endgültige Einreichung ausgewählt. Letzteres wurde zum Siegermodell.
4-7.Pipeline
Entschuldigen Sie die PowerPoint-Folie, aber ich habe sie schließlich mit der folgenden Pipeline abgeschlossen.
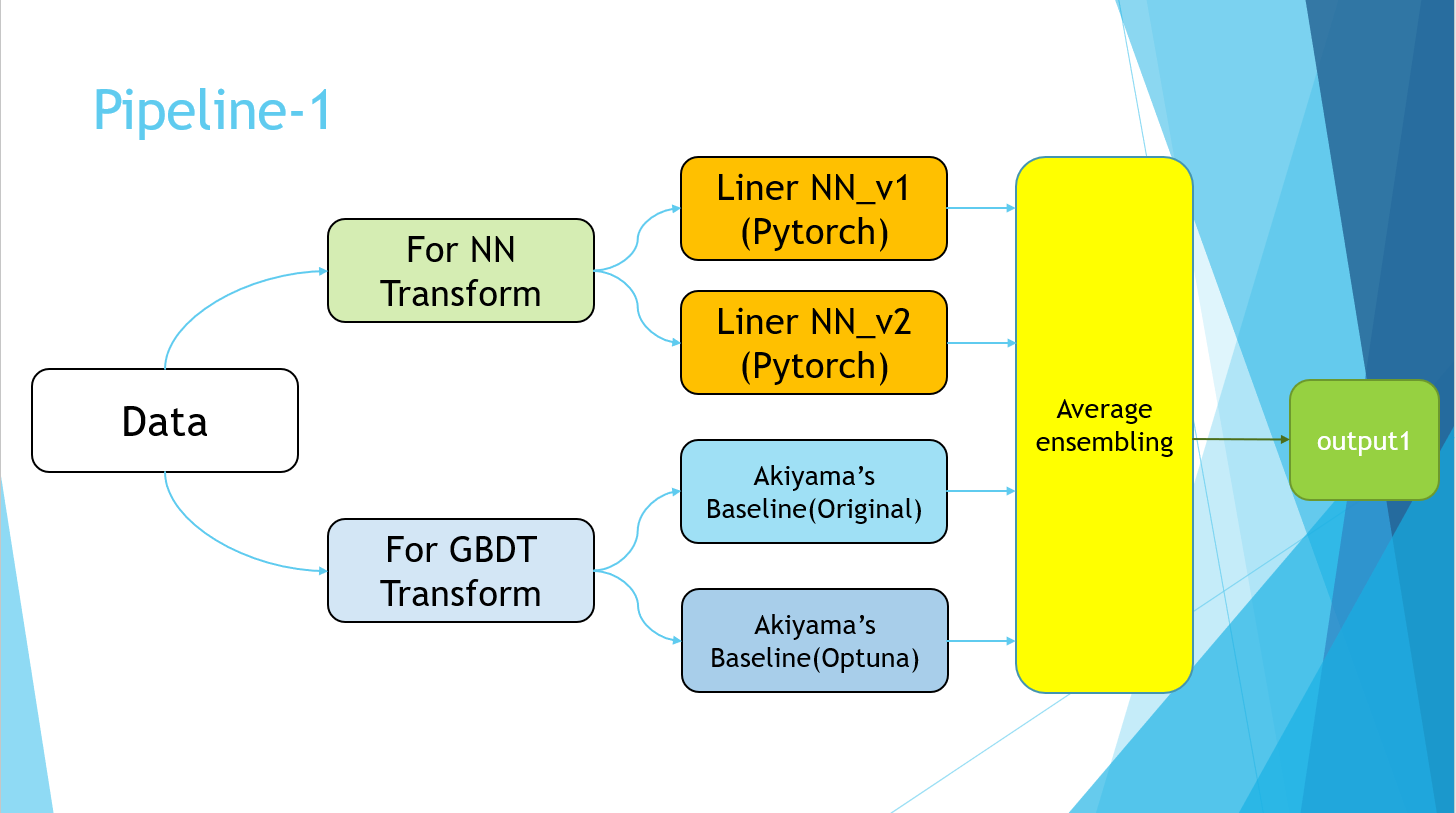
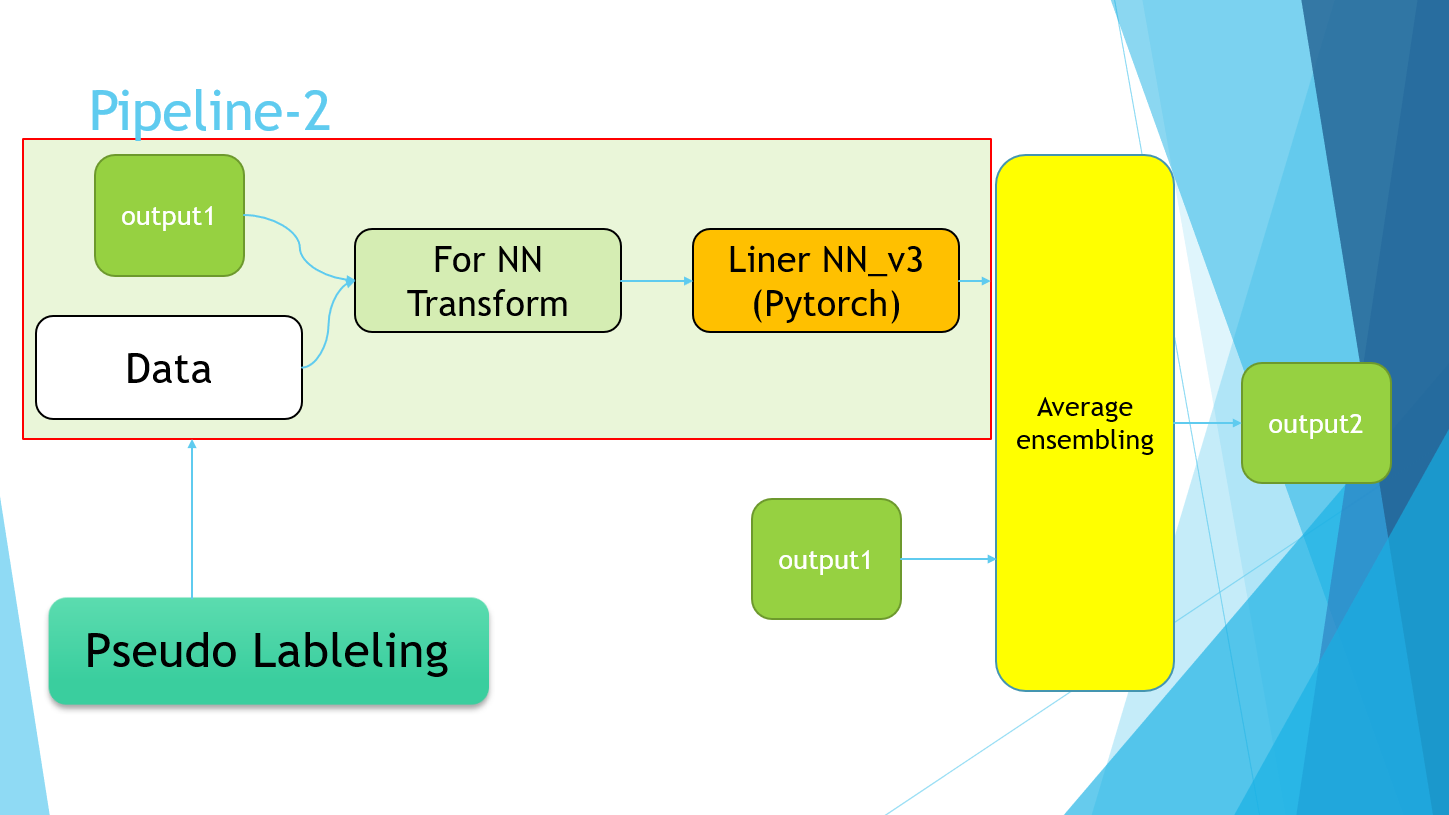
Der Pipeline-2-Teil bezieht sich einfach auf die Implementierung der Pseudo-Kennzeichnung. Schließlich wurden die Durchschnittswerte der vier Modelle von Pipeline-1 und der Modelle, die unter Verwendung der Pseudo-Markierungsdaten von Pipeline-2 trainiert wurden, genommen.
5. Ergebnis
Gewonnen. (Hast du jemals die Meisterschaft in deinem Leben gewonnen ...?) Was die Methode betrifft, habe ich das Gefühl, dass ich sie perfekt gemacht habe, und ich habe das Gefühl, etwas erreicht zu haben. Am zweiten Tag war ich die ganze Zeit auf dem ersten Platz und hatte Angst, nicht überholt zu werden, also war ich eher erleichtert als glücklich ...
6. Überlegung
Da dies ein privater Wettbewerb ist, werde ich es unterlassen, die Lösung anderer Leute zur Sprache zu bringen. Eine andere Sache, die ich versuchen wollte, ist von oben ・ Erstellung der Merkmalsmenge ・ Katzenschub ・ Hyperparameteranpassung des NN-Modells Es ist wie es ist. Wenn tatsächlich ein Wettbewerb mit diesen Daten zu Kaggle stattfinden würde, würde meine Punktzahl unter der Bronzemedaille liegen, daher denke ich, dass noch Verbesserungspotenzial besteht.
7. Am Ende
Vielen Dank an alle Organisatoren, einschließlich der AI & Machine Learning Society (AIMS) der Universität Osaka, die Moderatoren am ersten Tag und die Teilnehmer des Wettbewerbs! Ich hoffe, meine Lösung ist so hilfreich wie möglich.
8. Referenzen
- Daisuke Kadowaki Takashi Sakata Keisuke Hosaka Yuji Hiramatsu gewinnt mit Kaggle Data Analysis Technology Technology Review Company 2019
Recommended Posts